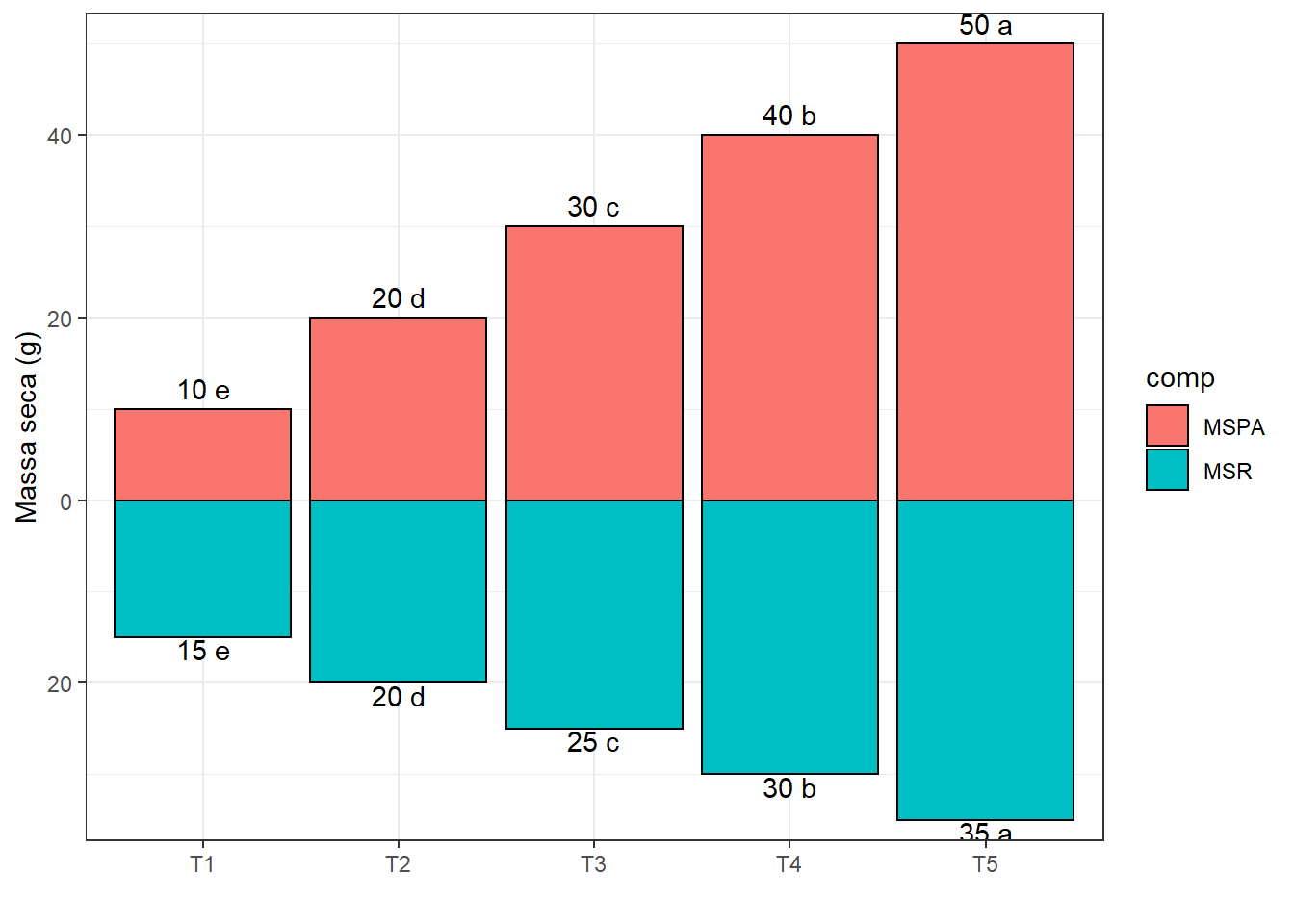2 Gráfico de Colunas
O gráfico em colunas consiste em construir retângulos, em que uma das dimensões é proporcional à magnitude a ser representada (\(n_i\) ou \(f_i\)), sendo a outra arbitrária, porém igual para todas as colunas. Essas colunas são dispostas paralelamente umas às outras de forma vertical.
Além do título e fonte de referências deve-se observar o seguinte:
- as colunas devem ter todas a mesma largura;
- a distância entre as colunas deve ser constante e de preferência menor que a largura das colunas.
2.1 Conjunto de dados
tratamentos=rep(c(paste("T",1:5)),e=4)
resposta=c(100,120,110,90,150,145,149,165,150,144,134,139,220,206,210,210,266,249,248,260)
## Média e Desvio-padrão (Por Tratamento)
media=tapply(resposta,tratamentos, mean)
desvio=tapply(resposta,tratamentos,sd)barplot(media)
2.2 Adicionando melhorias
barplot(media,
las=1,
col="lightyellow",
ylab="Resposta",
xlab="Tratamentos",
ylim=c(0,300))
abline(h=0)
Comandos:
las=1: deixar escala do eixo Y na vertical
col=“cor”: mudar cor das barras (Ex. “red,”“blue,”“green” ou gray.colors(quantidade de tonalidades) para escala cinza ou rainbow(quantidade de cores) para escala colorida. Também é possível específicar a cor de cada barra (col=c(“red,”“green,”“yellow,”“gray,”“blue”))).
xlab e ylab: nomear eixo X e Y
xlim e ylim: escala do eixo X e Y
abline(h=0): linha na horizontal em Y=0 (No caso de vertical, abline(v=0)). É possível alterar a cor pela função “col=”cor"" e o tracejado pelo “lty=número” (Ver o Help do comando)
2.3 Barras de desvio-padrão
bar=barplot(media,
las=1,
col="lightyellow",
ylab="Resposta",
xlab="Tratamentos",
ylim=c(0,300))
abline(h=0)
arrows(bar,media+desvio,bar,media-desvio,length = 0.1,angle=90,code=3)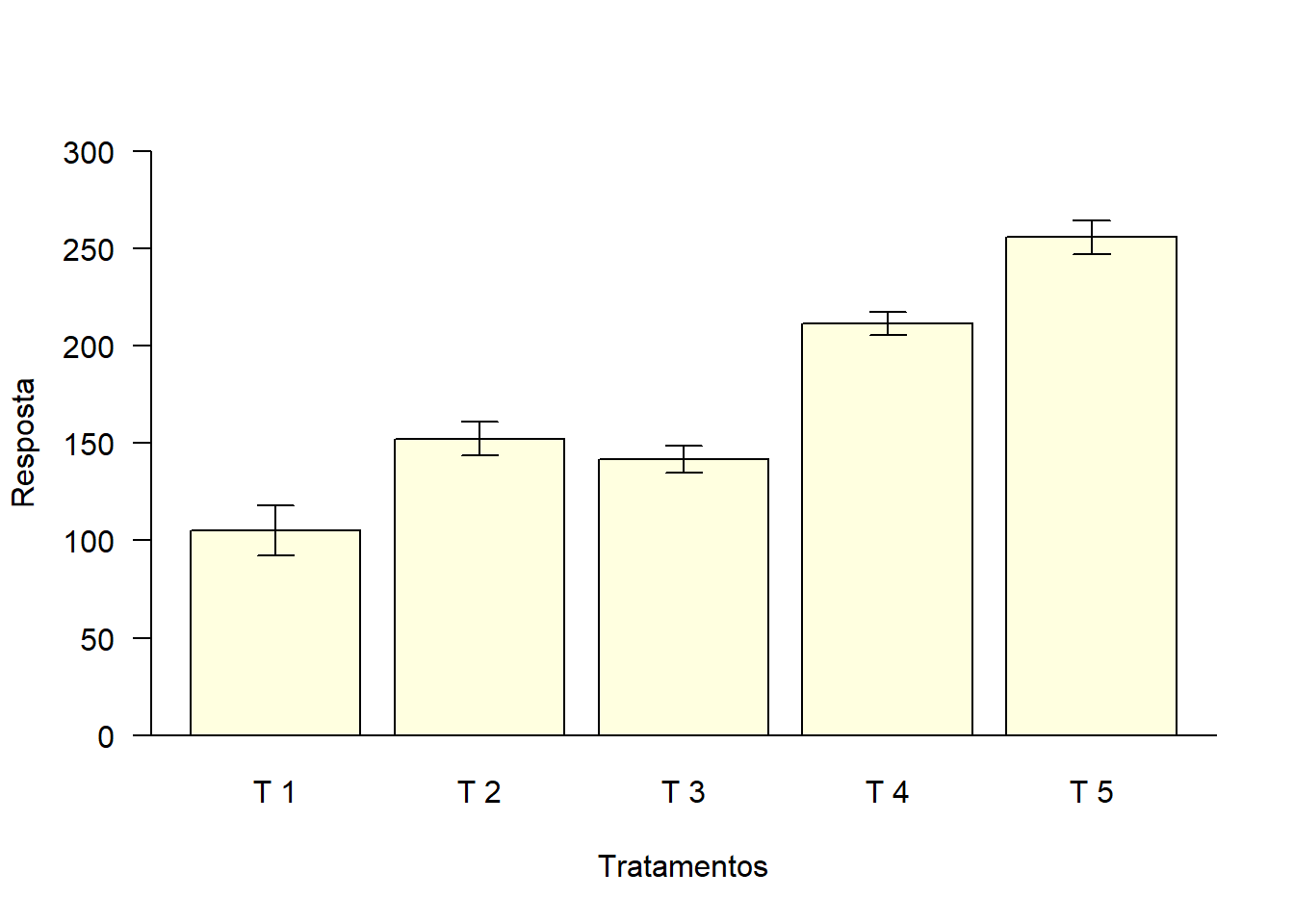
2.4 Unidade do eixo Y
(Ex. \(Kg\ ha^{-1}\))
bar=barplot(media,
las=1,
col="lightyellow",
ylab=expression("Resposta"*" "*(kg*" "*ha^-1)),
xlab="Tratamentos",
ylim=c(0,300))
abline(h=0)
arrows(bar,media+desvio,bar,media-desvio,length = 0.1,angle=90,code=3)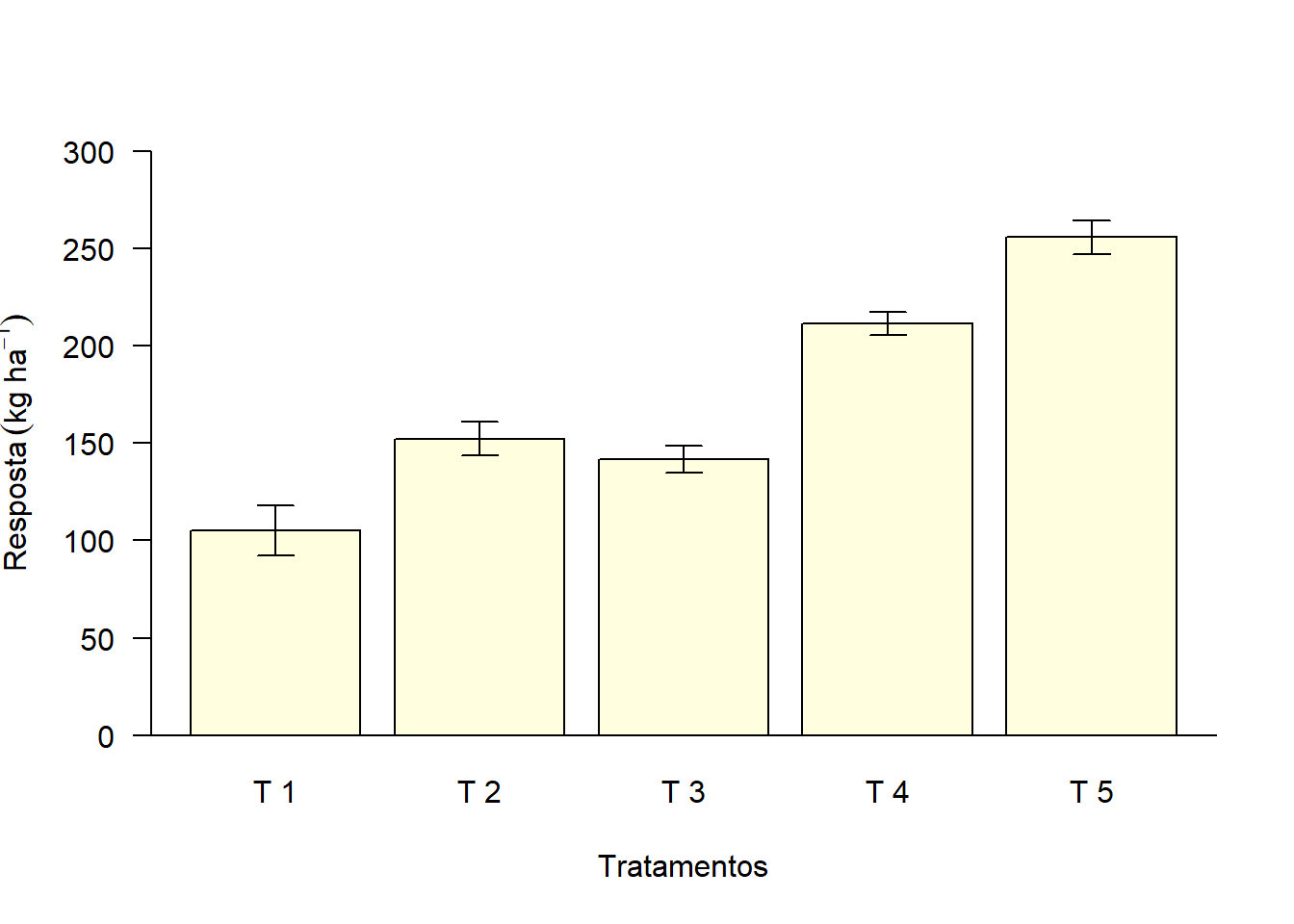
2.5 Média dos tratamentos
bar=barplot(media,
las=1,
col="lightyellow",
ylab=expression("Resposta"*" "*(kg*" "*ha^-1)),
xlab="Tratamentos",
ylim=c(0,300))
abline(h=0)
text(bar,media+desvio+10,media)
arrows(bar,media+desvio,bar,media-desvio,length = 0.1,angle=90,code=3)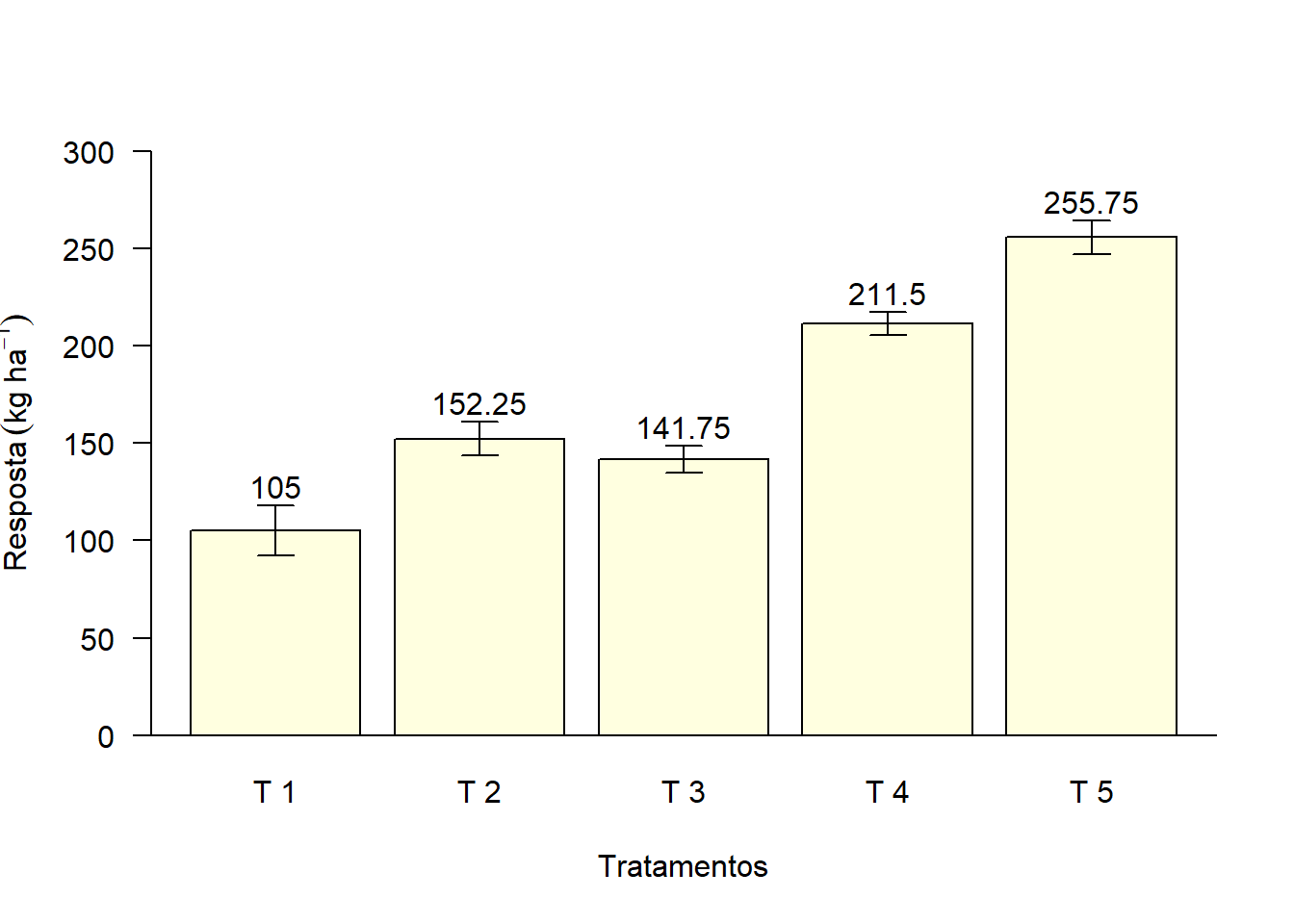
2.6 Separação de casa decimal
options(OutDec=",")
bar=barplot(media,
las=1,
col="lightyellow",
ylab=expression("Resposta"~(kg~ha^-1)),
xlab="Tratamentos",
ylim=c(0,300))
abline(h=0)
text(bar,media+desvio+10,media)
arrows(bar,media+desvio,bar,media-desvio,length = 0.1,angle=90,code=3)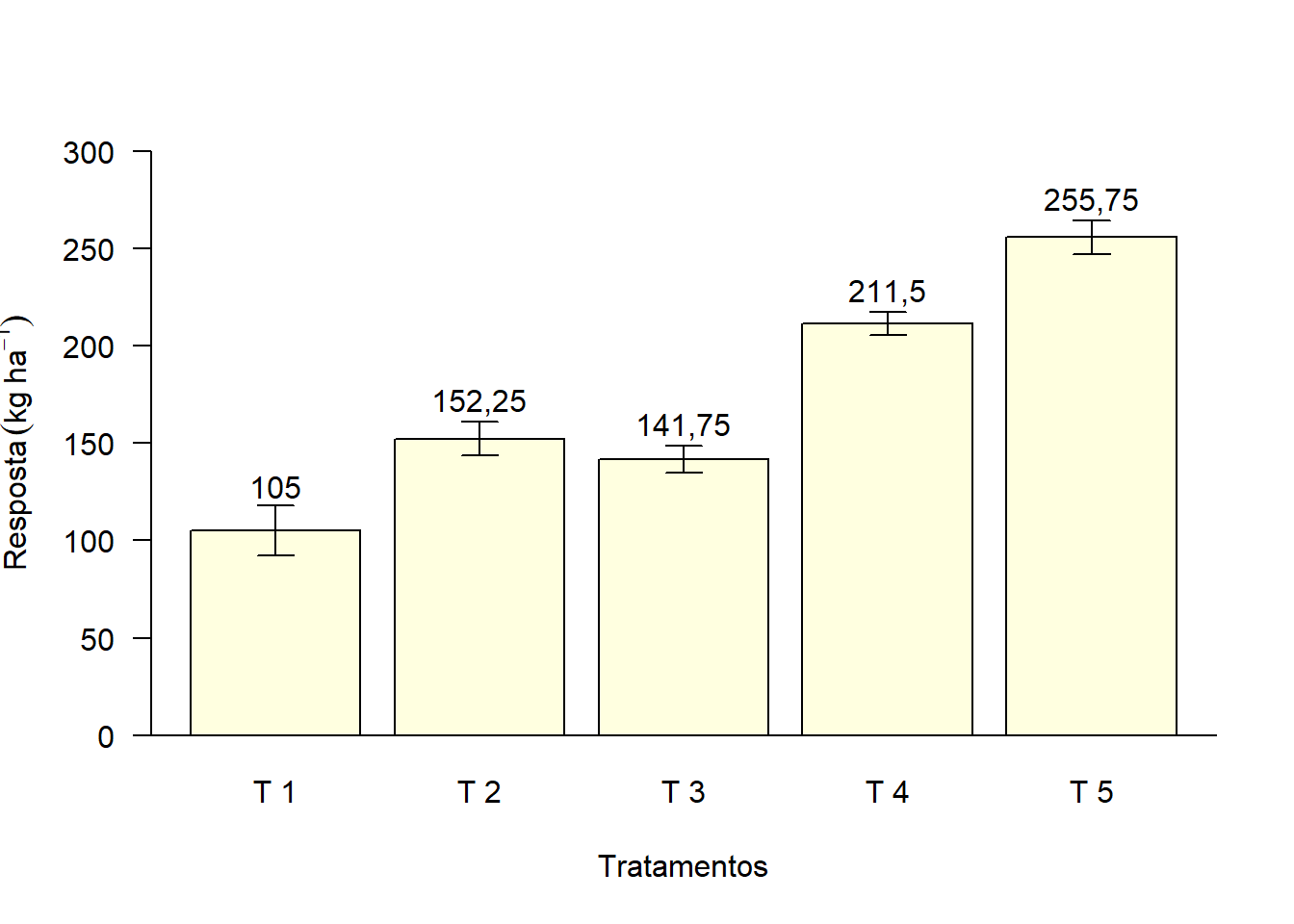
2.7 Letras do teste de comparação
tukey=c("d","c","c","b","a")
options(OutDec=",")
bar=barplot(media,
las=1,
col="lightyellow",
ylab=expression("Resposta"~(kg~ha^-1)),
xlab="Tratamentos",
ylim=c(0,300))
abline(h=0)
text(bar,media+desvio+10,paste(round(media,0),tukey))
arrows(bar,media+desvio,bar,media-desvio,length = 0.1,angle=90,code=3)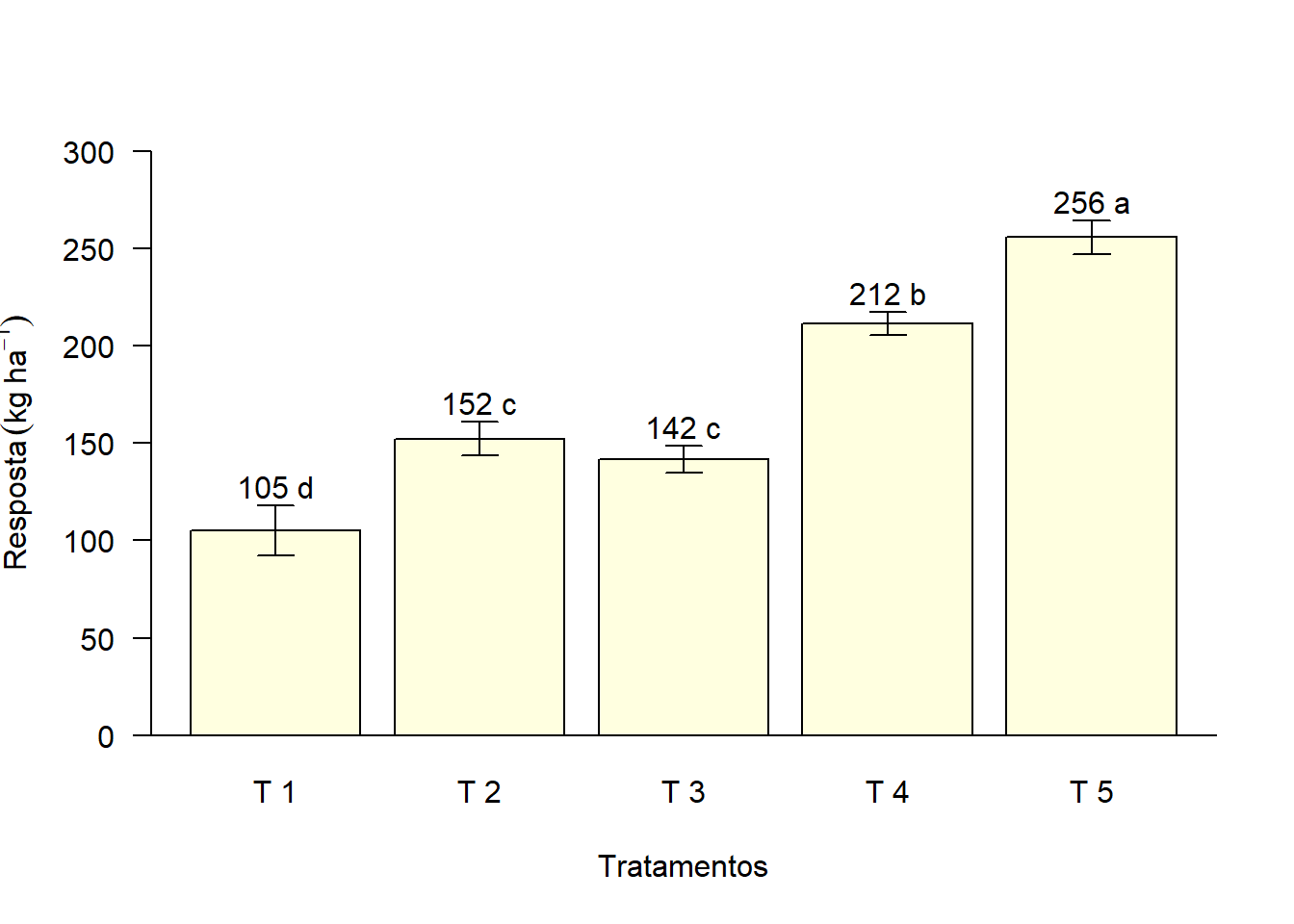
2.8 Pacote Agricolae
2.8.1 Conjunto de dados
tratamentos=rep(c(paste("T",1:5)),e=4)
resposta=c(100,120,110,90,150,145,149,165,150,144,134,139,220,206,210,210,266,249,248,260)2.8.2 Modelo de Anova
modelo=aov(resposta~tratamentos)library(agricolae)
a=HSD.test(modelo,"tratamentos", group = T)2.8.3 Gráfico com média
plot(a, las=1)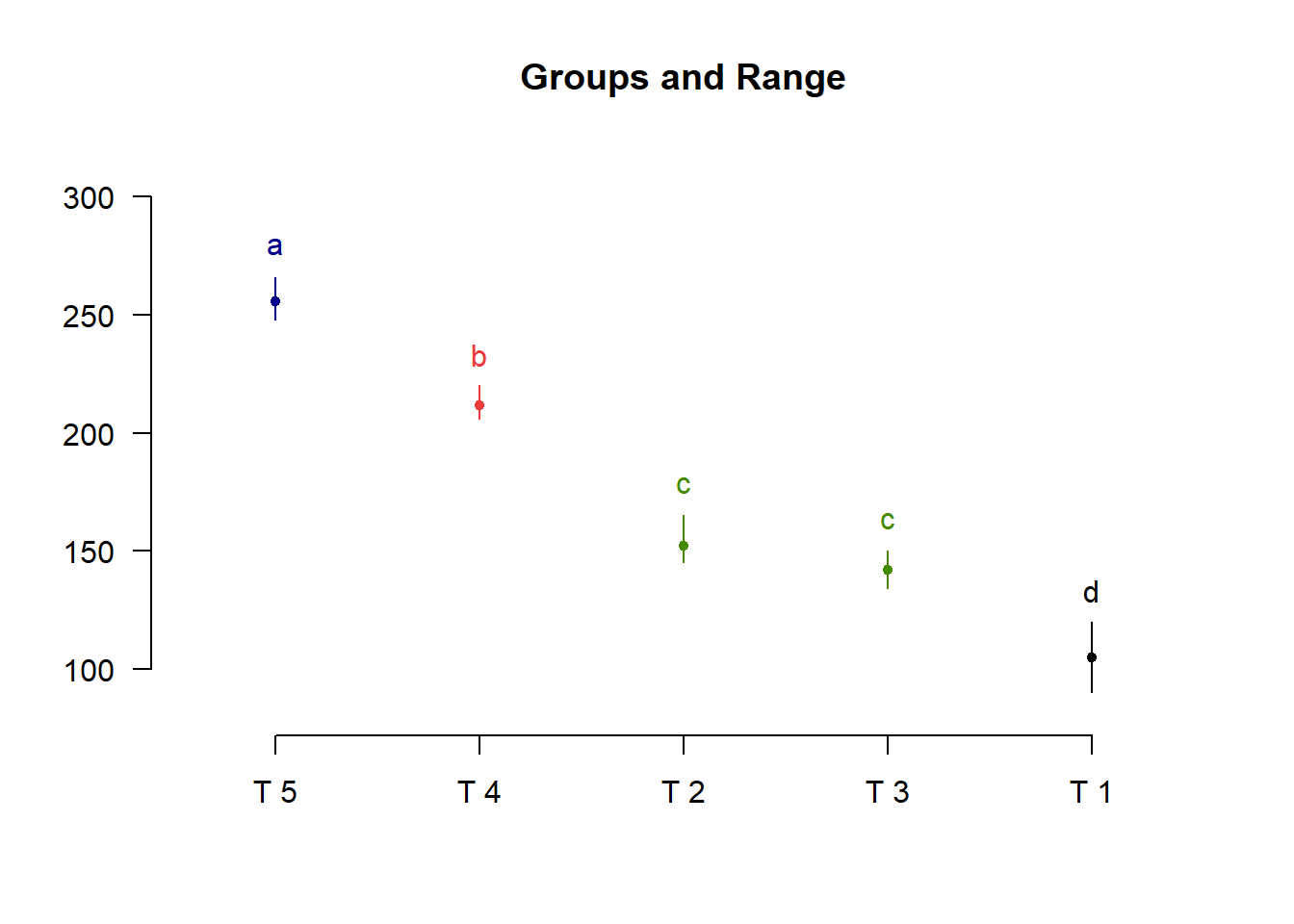
2.8.4 Gráfico de barras
bar.group(a$groups,
col="lightblue",
las=1,
ylim=c(0,300))
abline(h=0)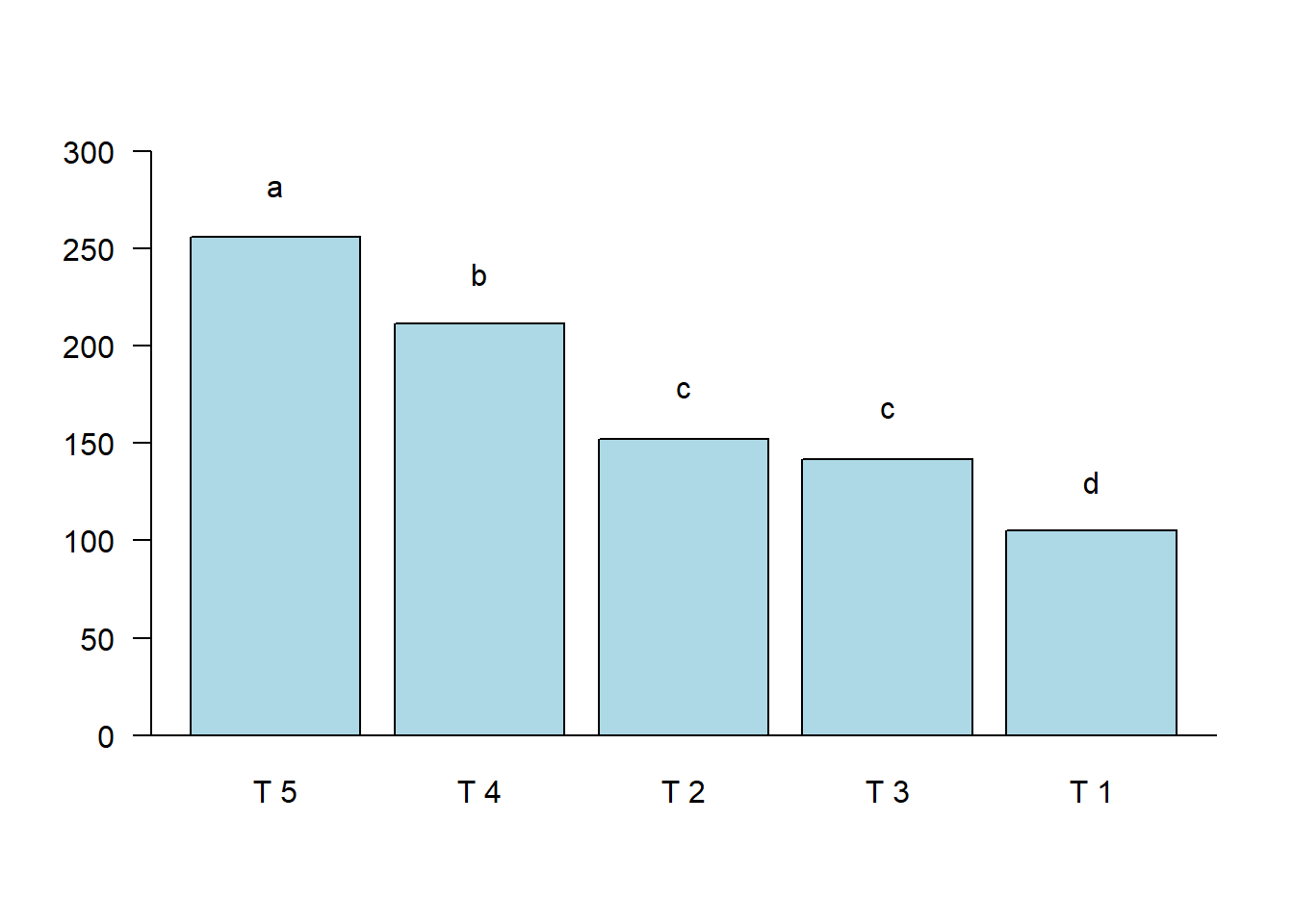
2.8.5 Barras de desvio-padrão
bar.err(a$means,
variation="SD",
col="lightblue",
las=1,
ylim=c(0,300))
abline(h=0)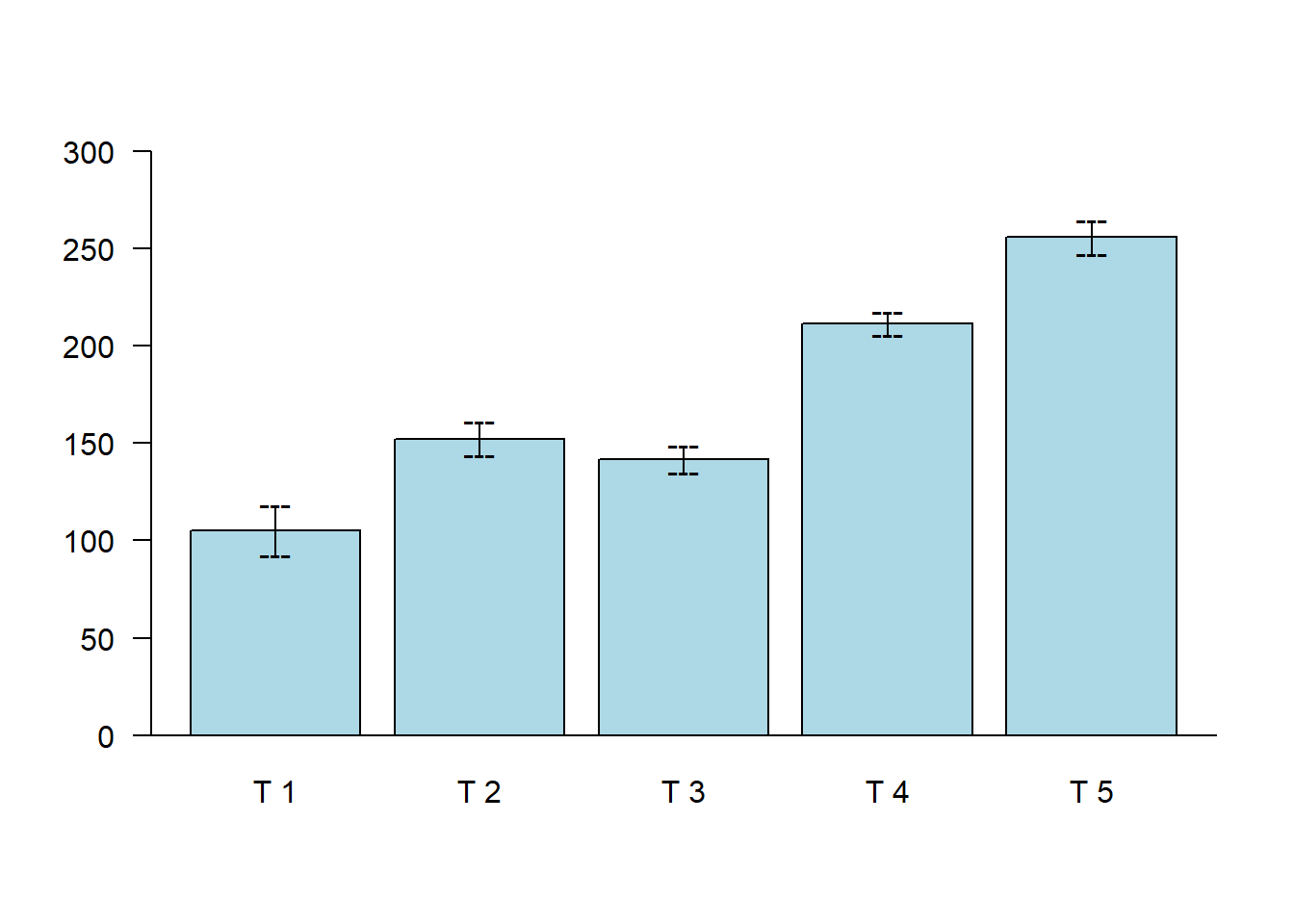
2.8.6 Barras de erro padrão
bar.err(a$means,
variation="SE",
col="lightblue",
las=1,
ylim=c(0,300))
abline(h=0)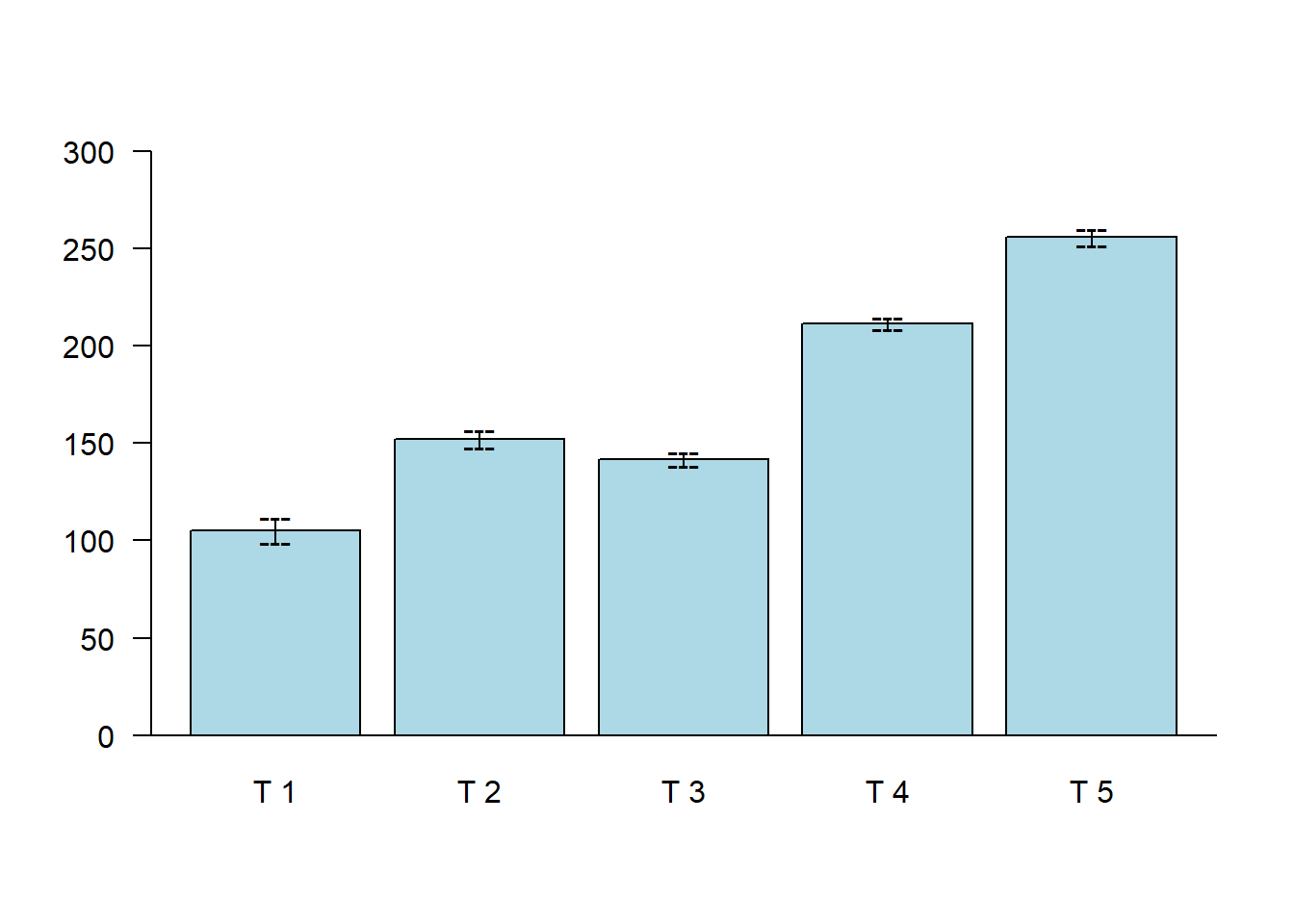
2.8.7 Barras de máximo-mínimo
bar.err(a$means,
variation="range",
col="lightblue",
las=1,
ylim=c(0,300))
abline(h=0)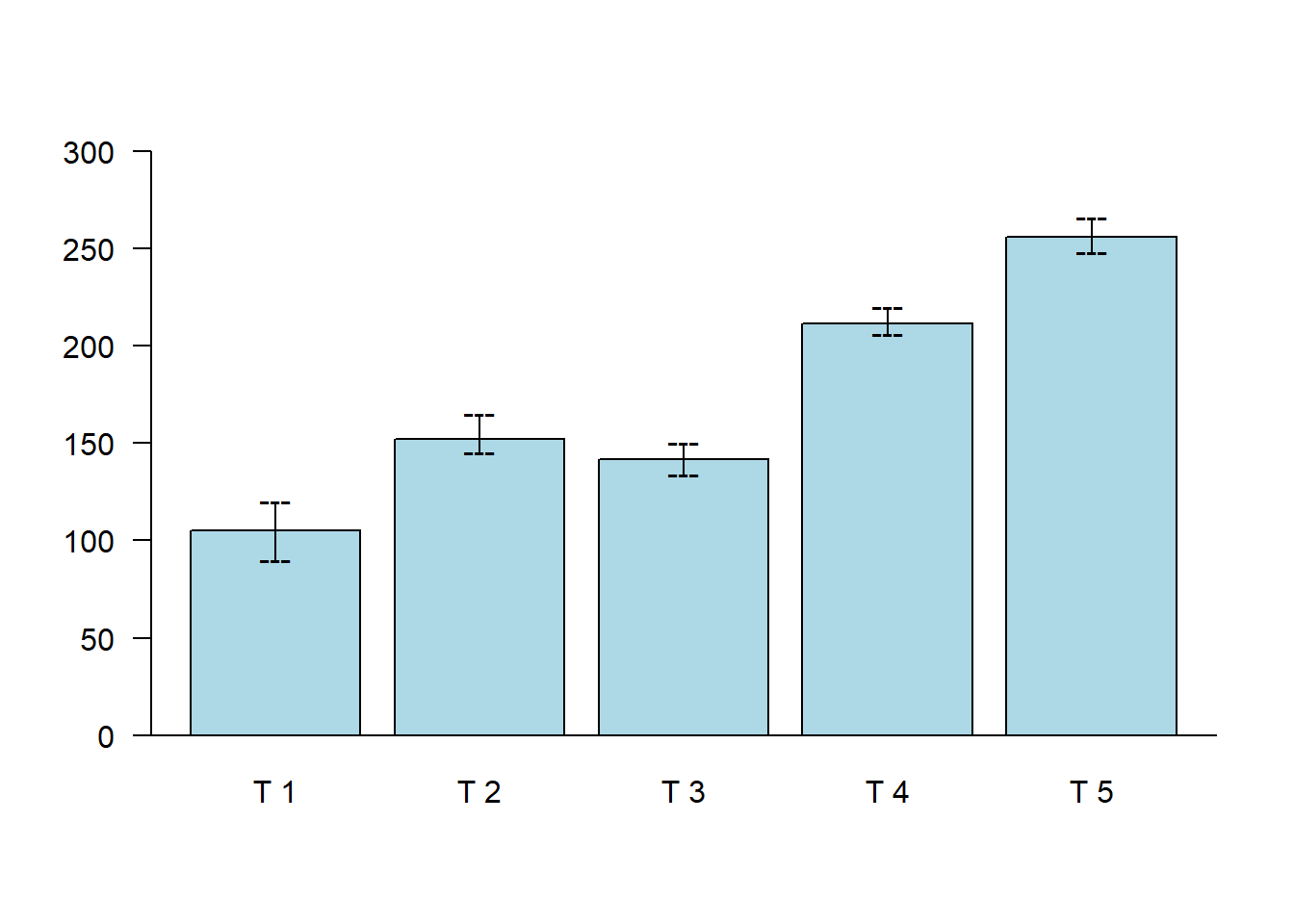
2.8.8 Barras da distância interquartil
bar.err(a$means,
variation="IQR",
col="lightblue",
las=1,
ylim=c(0,300))
abline(h=0)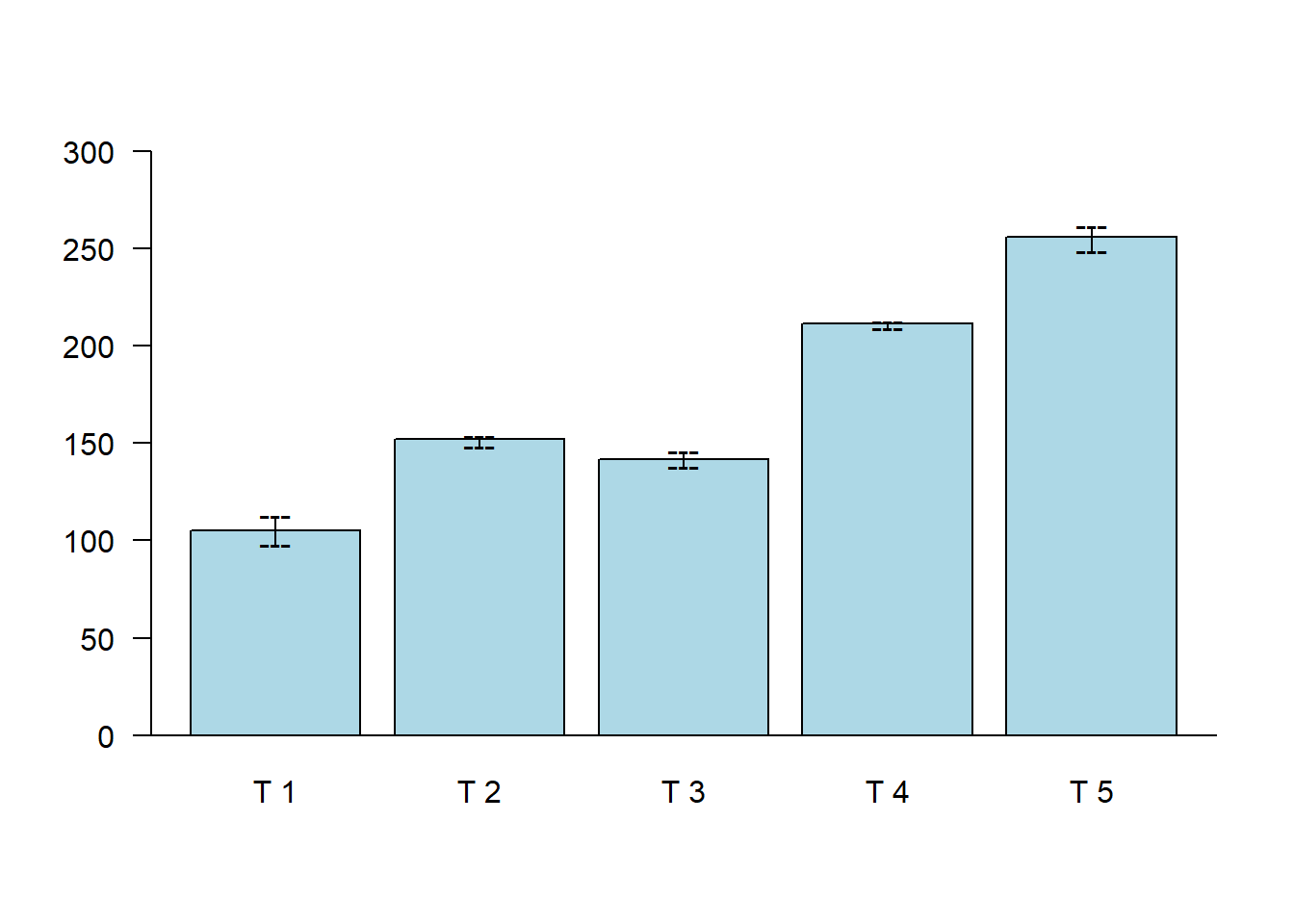
2.9 Pacote ggplot2 e ggpubr
2.9.1 Conjunto de dados
Vamos trabalhar com três experimentos em DIC com quatro tratamentos e três repetições cada.
exp1=c(10,12,13,18,19,16,5,6,5,25,26,28)
exp2=c(9,12,11,18,20,16,7,6,9,25,28,28)
exp3=c(9,12,13,18,22,15,3,6,4,25,30,28)
Trat=rep(c(paste("T",1:4)),e=3)
dados=data.frame(Trat,exp1,exp2,exp3)
dados$Trat=as.factor(Trat)Obs. Para facilitar, vamos realizar a análise direto pelo pacote ExpDes.pt (é necessário instalar o pacote)
2.9.2 Análise de exp1
ExpDes.pt::dic(Trat,exp1)## ------------------------------------------------------------------------
## Quadro da analise de variancia
## ------------------------------------------------------------------------
## GL SQ QM Fc Pr>Fc
## Tratamento 3 719,58 239,861 130,83 3,8864e-07
## Residuo 8 14,67 1,833
## Total 11 734,25
## ------------------------------------------------------------------------
## CV = 8,88 %
##
## ------------------------------------------------------------------------
## Teste de normalidade dos residuos ( Shapiro-Wilk )
## Valor-p: 0,3563889
## De acordo com o teste de Shapiro-Wilk a 5% de significancia, os residuos podem ser considerados normais.
## ------------------------------------------------------------------------
##
## ------------------------------------------------------------------------
## Teste de homogeneidade de variancia
## valor-p: 0,6539247
## De acordo com o teste de bartlett a 5% de significancia, as variancias podem ser consideradas homogeneas.
## ------------------------------------------------------------------------
##
## Teste de Tukey
## ------------------------------------------------------------------------
## Grupos Tratamentos Medias
## a T 4 26,33333
## b T 2 17,66667
## c T 1 11,66667
## d T 3 5,333333
## ------------------------------------------------------------------------2.9.3 Análise de exp2
ExpDes.pt::dic(Trat,exp2)## ------------------------------------------------------------------------
## Quadro da analise de variancia
## ------------------------------------------------------------------------
## GL SQ QM Fc Pr>Fc
## Tratamento 3 684,92 228,306 78,276 2,8606e-06
## Residuo 8 23,33 2,917
## Total 11 708,25
## ------------------------------------------------------------------------
## CV = 10,84 %
##
## ------------------------------------------------------------------------
## Teste de normalidade dos residuos ( Shapiro-Wilk )
## Valor-p: 0,2365244
## De acordo com o teste de Shapiro-Wilk a 5% de significancia, os residuos podem ser considerados normais.
## ------------------------------------------------------------------------
##
## ------------------------------------------------------------------------
## Teste de homogeneidade de variancia
## valor-p: 0,9823917
## De acordo com o teste de bartlett a 5% de significancia, as variancias podem ser consideradas homogeneas.
## ------------------------------------------------------------------------
##
## Teste de Tukey
## ------------------------------------------------------------------------
## Grupos Tratamentos Medias
## a T 4 27
## b T 2 18
## c T 1 10,66667
## c T 3 7,333333
## ------------------------------------------------------------------------2.9.4 Análise de exp3
ExpDes.pt::dic(Trat,exp3)## ------------------------------------------------------------------------
## Quadro da analise de variancia
## ------------------------------------------------------------------------
## GL SQ QM Fc Pr>Fc
## Tratamento 3 894,25 298,083 47,066 1,9902e-05
## Residuo 8 50,67 6,333
## Total 11 944,92
## ------------------------------------------------------------------------
## CV = 16,32 %
##
## ------------------------------------------------------------------------
## Teste de normalidade dos residuos ( Shapiro-Wilk )
## Valor-p: 0,9419794
## De acordo com o teste de Shapiro-Wilk a 5% de significancia, os residuos podem ser considerados normais.
## ------------------------------------------------------------------------
##
## ------------------------------------------------------------------------
## Teste de homogeneidade de variancia
## valor-p: 0,7583526
## De acordo com o teste de bartlett a 5% de significancia, as variancias podem ser consideradas homogeneas.
## ------------------------------------------------------------------------
##
## Teste de Tukey
## ------------------------------------------------------------------------
## Grupos Tratamentos Medias
## a T 4 27,66667
## b T 2 18,33333
## c T 1 11,33333
## d T 3 4,333333
## ------------------------------------------------------------------------2.10 Utilizando o ggplot2
library(ggplot2)
library(gridExtra)2.10.1 Média e desvio-padrão
media=tapply(exp1, Trat, mean)
desvio=tapply(exp1, Trat, sd)
## Construindo uma nova data.frame com a media e desvio
dados1=data.frame(Trat=rownames(media),media,desvio)2.10.2 Gráfico básico
ggplot(dados1,
aes(x=Trat,y=media))+
geom_col()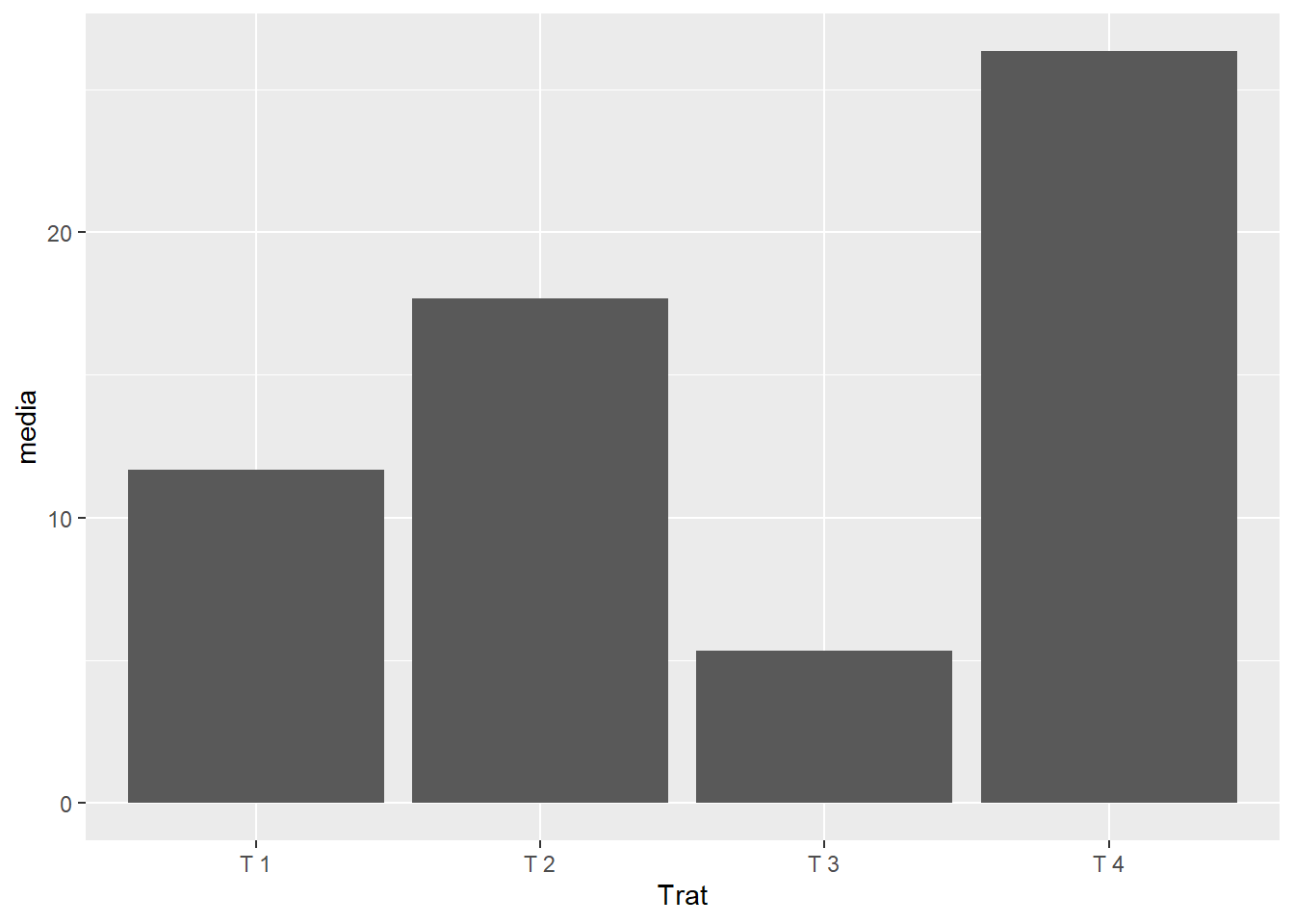
2.10.3 Média no gráfico
ggplot(dados1,
aes(x=Trat,y=media))+
geom_col()+
geom_text(label=round(media,1), vjust=-1) 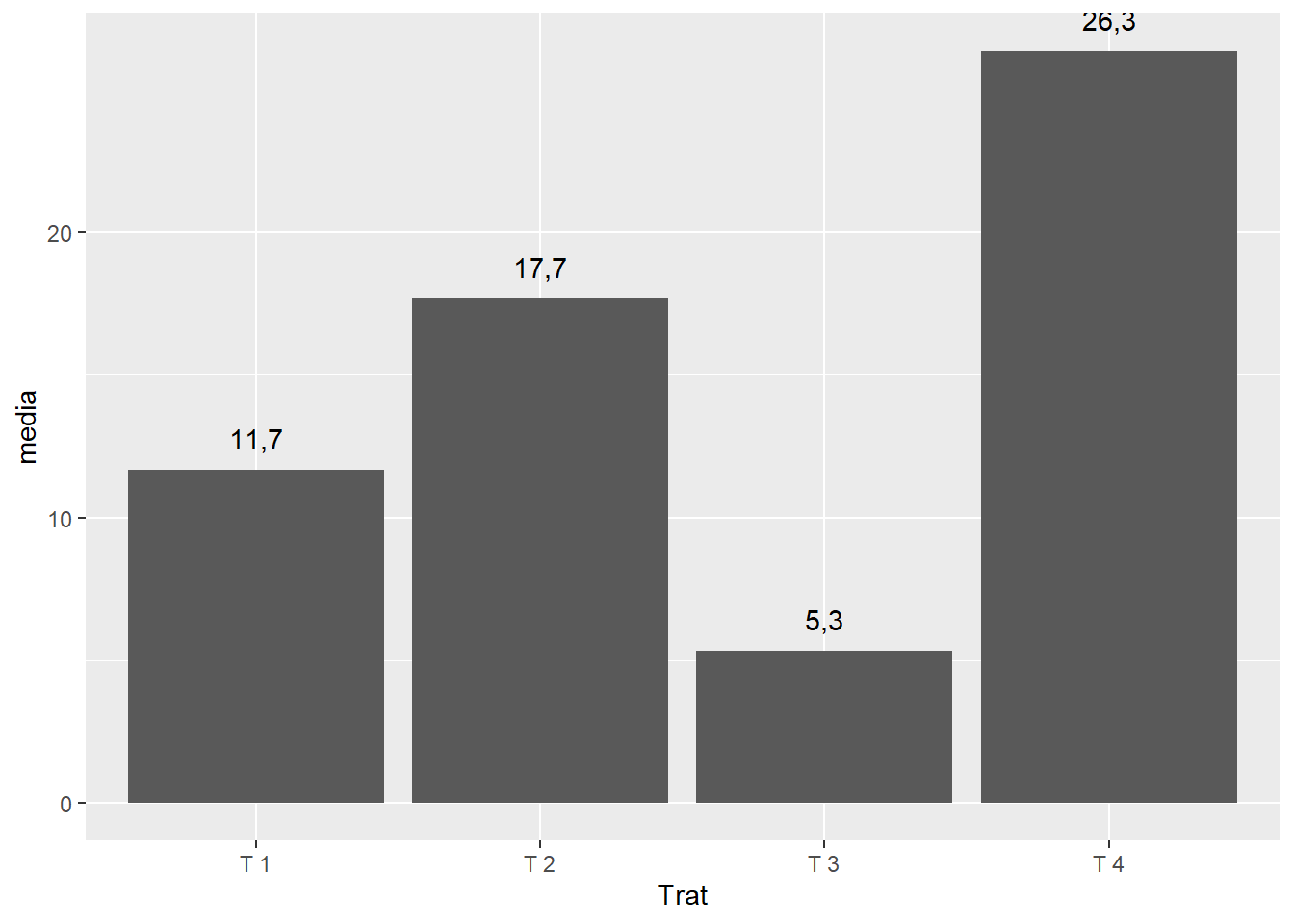
# Obs. Round é para arrendondar o valor, neste caso estamos pedindo até a primeira casa decimal2.10.4 Letras do teste de comparação
ggplot(dados1,
aes(x=Trat,y=media))+
geom_col()+
geom_text(label=paste(round(media,1),c("c","b","d","a")), vjust=-1)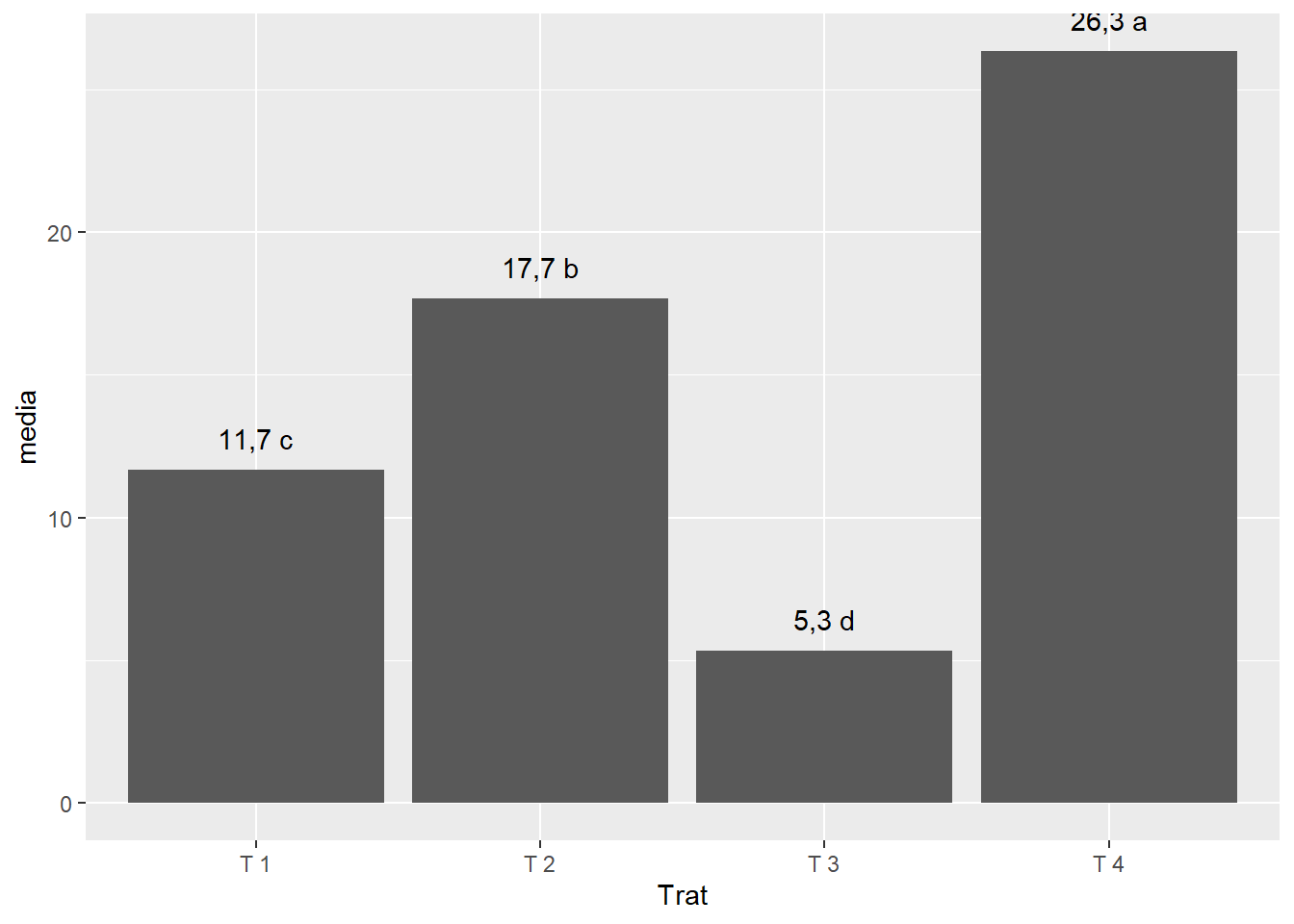
#Obs. a função paste serve para juntar palavras, nesse caso está juntando cada média com suas respectivas letras do teste de comparação de médias2.10.5 Escala do eixo Y
ggplot(dados1,
aes(x=Trat,y=media))+
geom_col()+
geom_text(label=paste(round(media,1),c("c","b","d","a")), vjust=-1)+
ylim(c(0,40))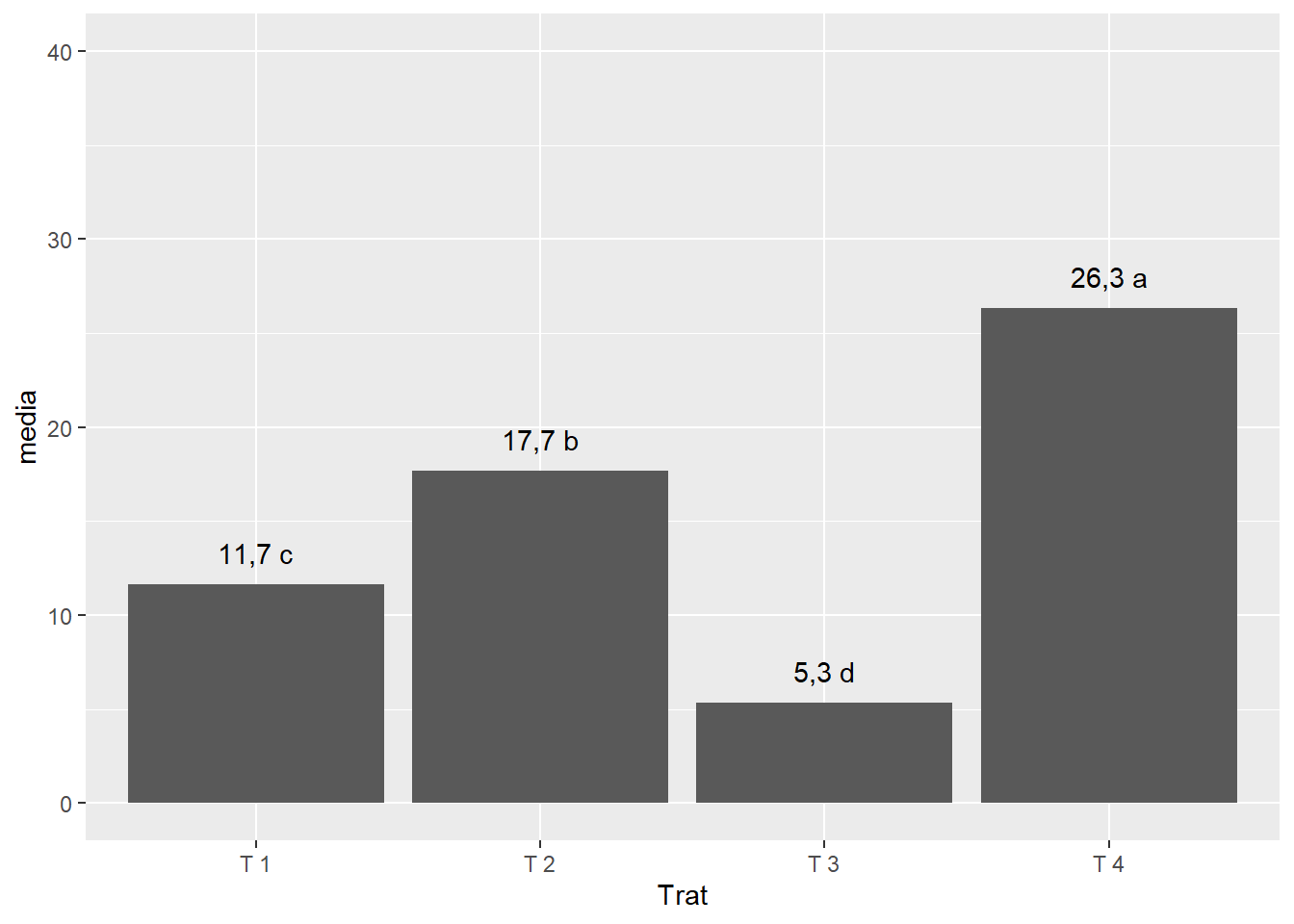
2.10.6 Cor das colunas
ggplot(dados1,
aes(x=Trat,y=media))+
geom_col(fill=c(1,2,3,4))+
geom_text(label=paste(round(media,1),c("c","b","d","a")), vjust=-1)+
ylim(c(0,40))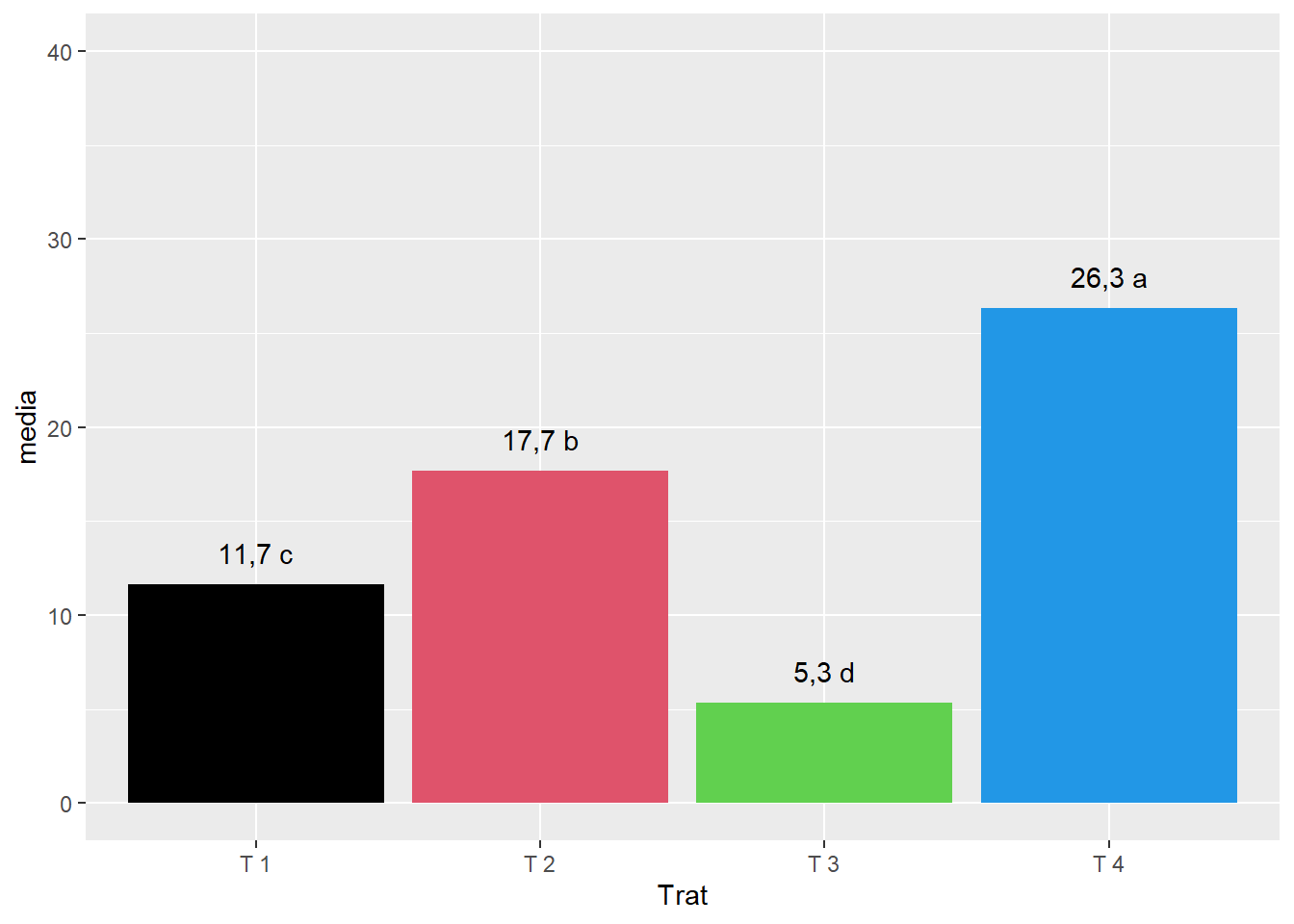
2.10.7 Removendo cor de fundo
ggplot(dados1,
aes(x=Trat,y=media))+
geom_col(fill=c(1,2,3,4))+
geom_text(label=paste(round(media,1),c("c","b","d","a")), vjust=-1)+
ylim(c(0,40))+
theme_bw()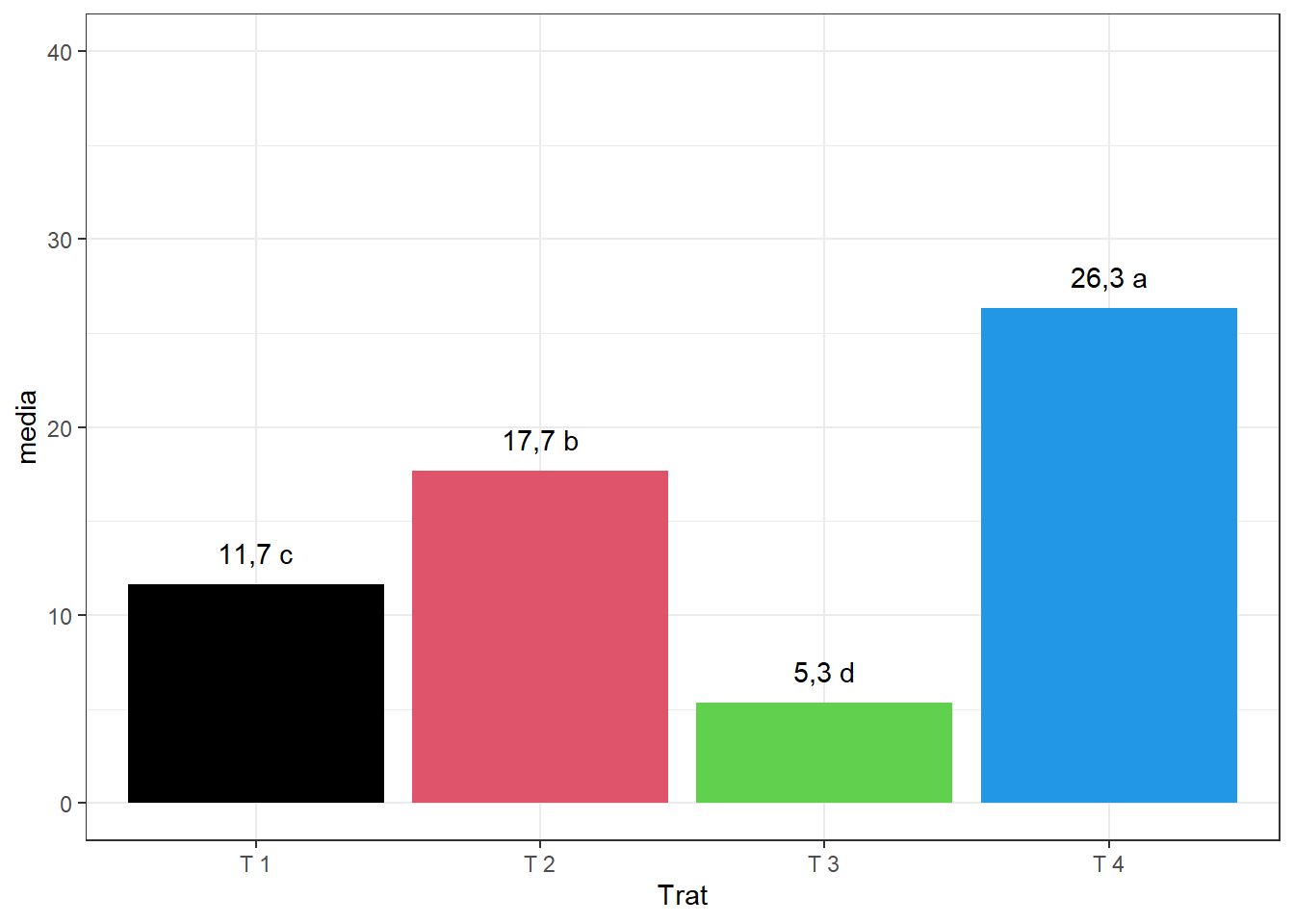
2.10.8 Removendo linhas de grade
ggplot(dados1,
aes(x=Trat,y=media))+
geom_col(fill=c(1,2,3,4))+
geom_text(label=paste(round(media,1),c("c","b","d","a")), vjust=-1)+
ylim(c(0,40))+
theme_bw()+
theme_classic()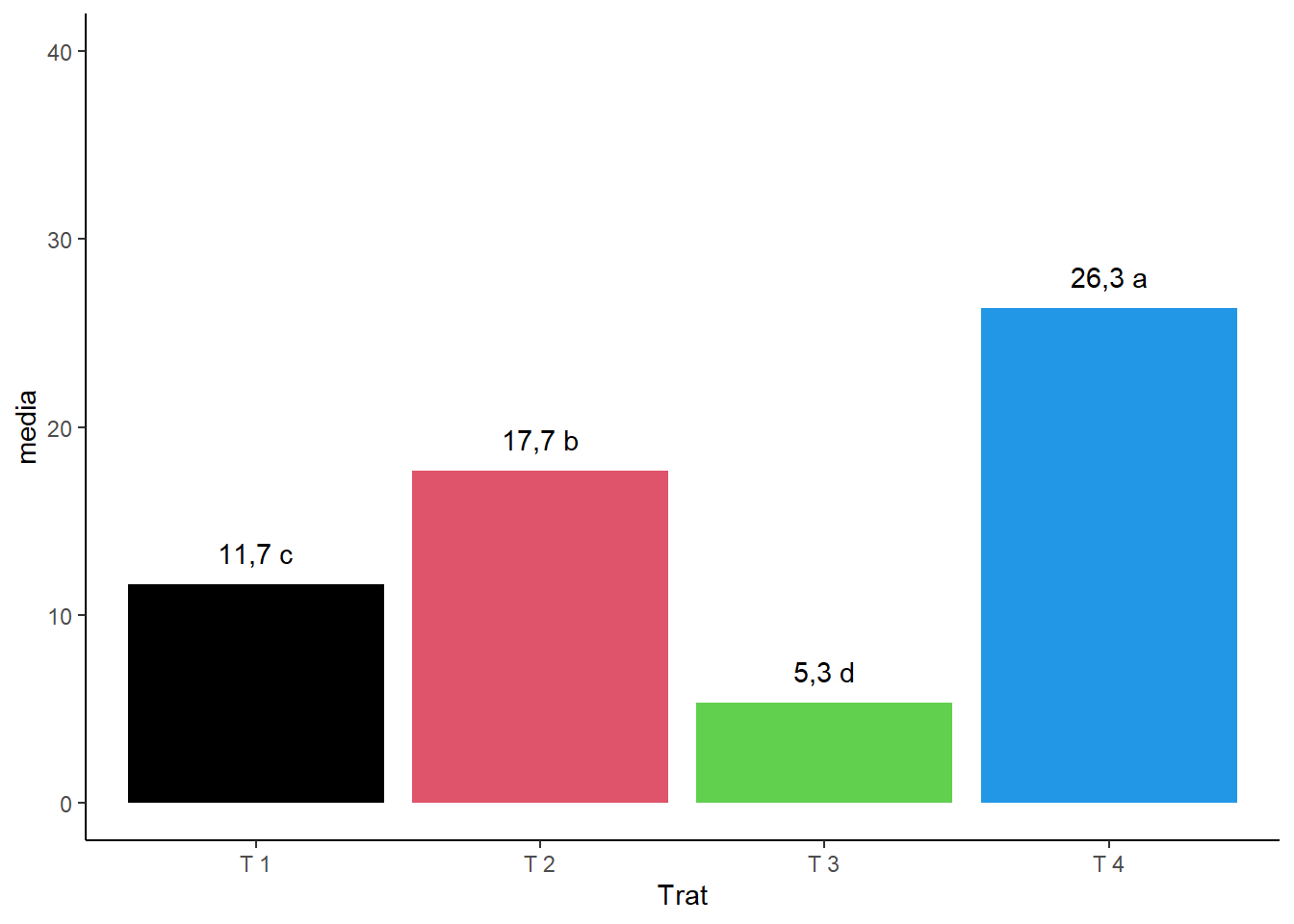
2.10.9 Nome dos eixos X e Y
Obs. A função expression() funciona nesses argumentos.
ggplot(dados1, aes(x=Trat,y=media))+
geom_col(fill=c(1,2,3,4))+
geom_text(label=paste(round(media,1),c("c","b","d","a")), vjust=-1)+
ylim(c(0,40))+
theme_bw()+
theme_classic()+
ylab("Resposta")+
xlab(" ")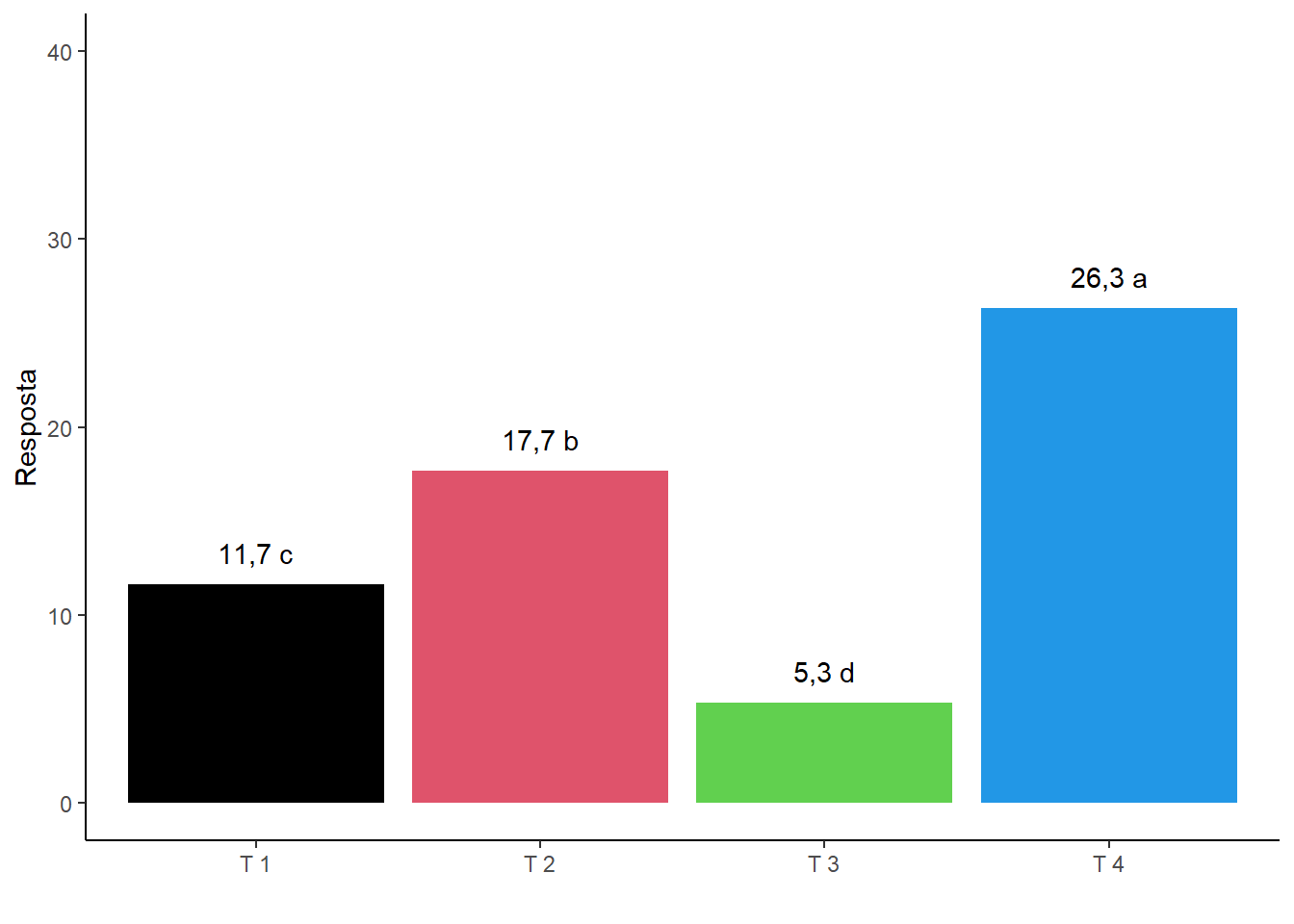
2.10.10 Cor do contorno das colunas
ggplot(dados1,
aes(x=Trat,y=media))+
geom_col(fill=c(1,2,3,4),col="black")+ # Modifiquei aqui
geom_text(label=paste(round(media,1),c("c","b","d","a")), vjust=-1)+
ylim(c(0,40))+
theme_bw()+
theme_classic()+
ylab("Resposta")+
xlab(" ")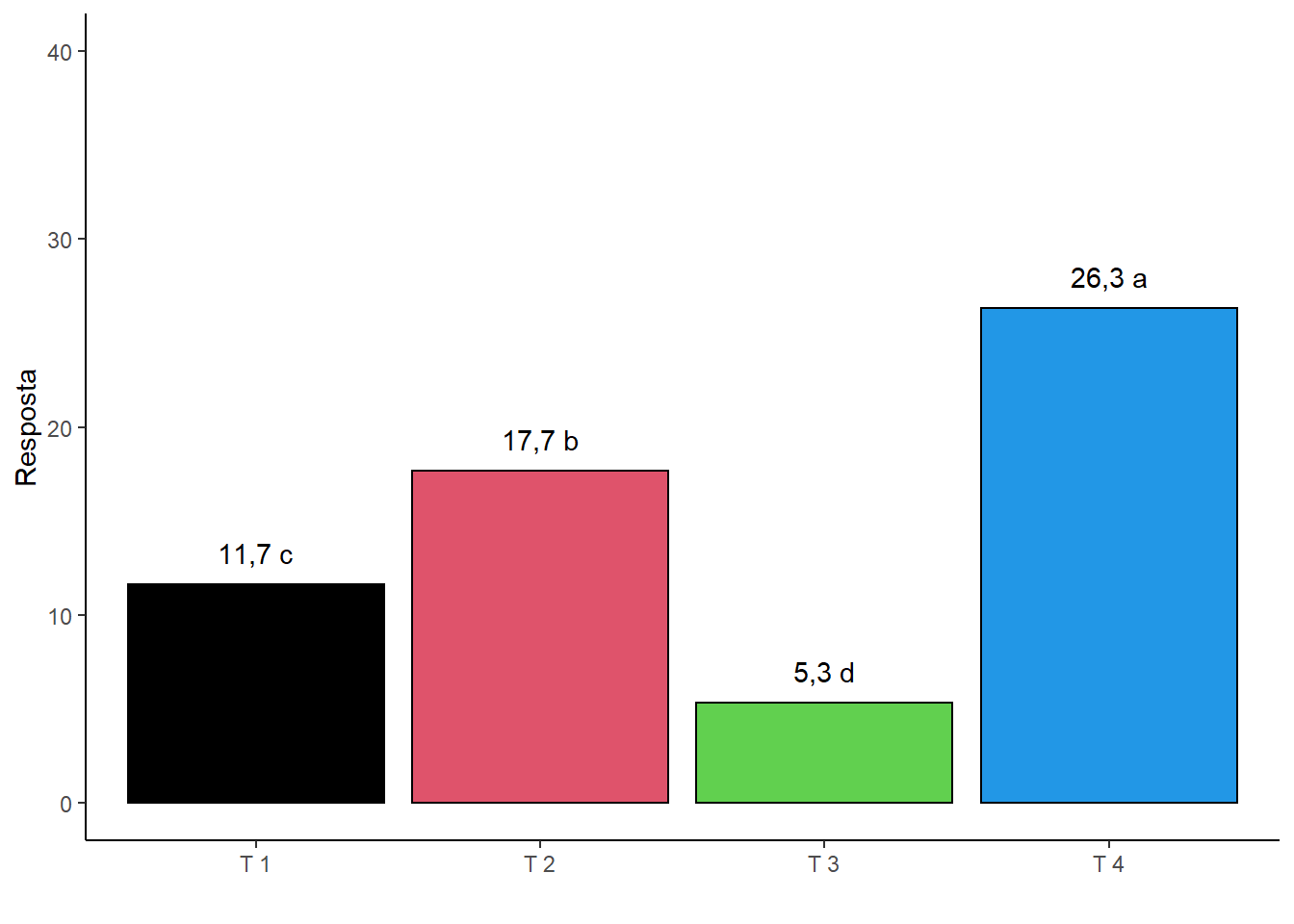
2.10.11 Barras de desvio-padrão
ggplot(dados1, aes(x=Trat,y=media))+
geom_col(fill=c(1,2,3,4),col="black")+
geom_text(label=paste(round(media,1),c("c","b","d","a")), vjust=-2)+
ylim(c(0,40))+
theme_bw()+
theme_classic()+
ylab("Resposta")+
xlab(" ")+
geom_errorbar(aes(ymax=media+desvio,ymin=media-desvio), width=0.25) # Width é a largura da barra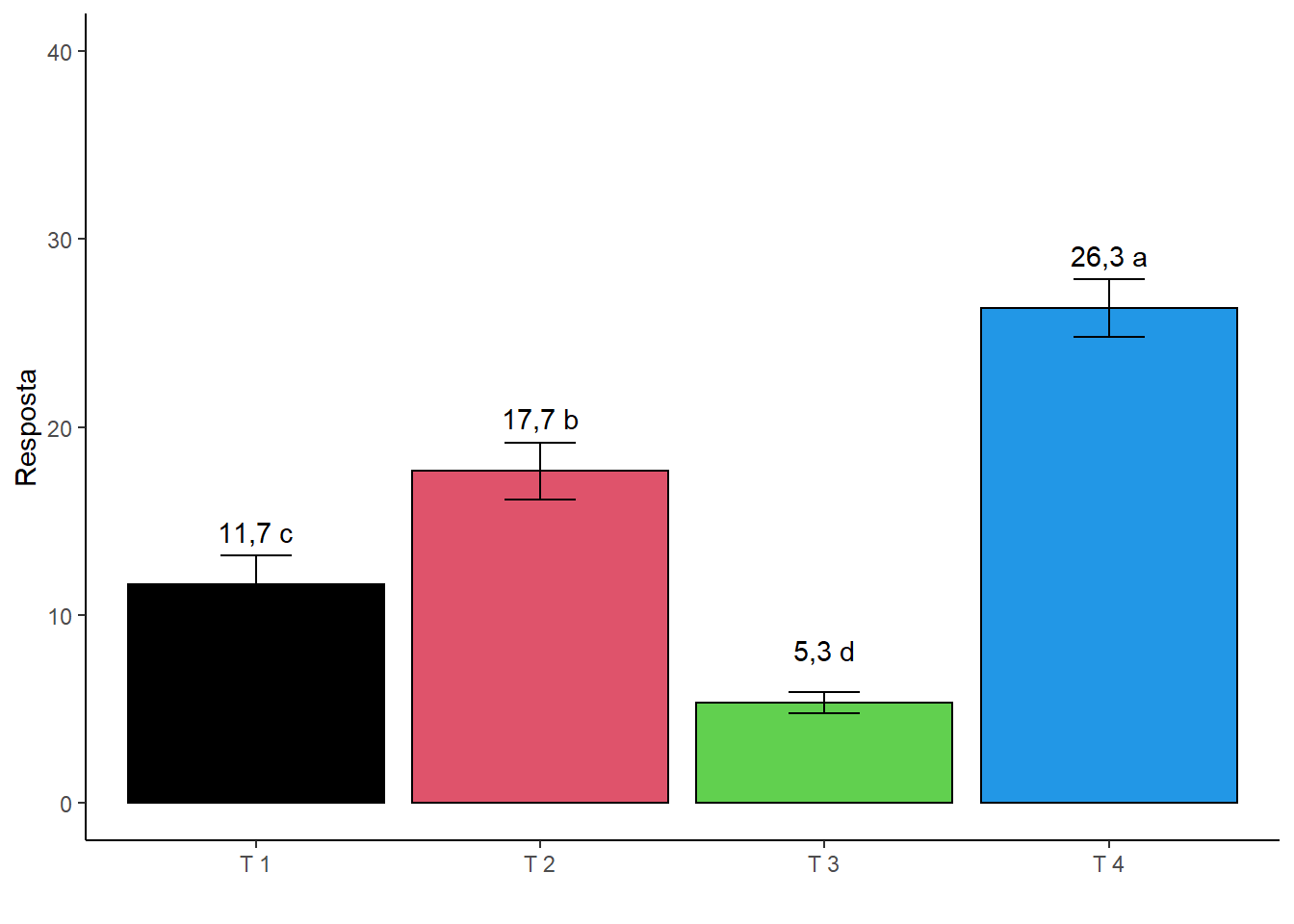
2.10.12 Juntando os gráficos
Obs. Vamos chamar todo o plot de cada uma das variáveis de a,b,c, respectivamente.
2.10.13 Variável exp1
media=tapply(exp1, Trat, mean)
desvio=tapply(exp1, Trat, sd)
dados1=data.frame(Trat=rownames(media),media,desvio)
a=ggplot(dados1, aes(x=Trat,y=media))+
geom_col(fill=c(1,2,3,4),col="black")+
geom_text(label=paste(round(media,1),
c("c","b","d","a")), vjust=-3)+
ylim(c(0,40))+theme_bw()+theme_classic()+ylab("Resposta")+xlab(" ")+
geom_errorbar(aes(ymax=media+desvio,
ymin=media-desvio), width=0.25)2.10.14 Variável exp2
media=tapply(exp2, Trat, mean)
desvio=tapply(exp2, Trat, sd)
dados2=data.frame(Trat=rownames(media),media,desvio)
b=ggplot(dados2, aes(x=Trat,y=media))+
geom_col(fill=c(1,2,3,4),col="black")+
geom_text(label=paste(round(media,1),
c("c","b","c","a")), vjust=-3)+
ylim(c(0,40))+theme_bw()+theme_classic()+ylab("Resposta")+xlab(" ")+
geom_errorbar(aes(ymax=media+desvio,
ymin=media-desvio), width=0.25)2.10.15 Variável exp3
media=tapply(exp3, Trat, mean)
desvio=tapply(exp3, Trat, sd)
dados3=data.frame(Trat=rownames(media),media,desvio)
c=ggplot(dados3, aes(x=Trat,y=media))+
geom_col(fill=c(1,2,3,4),col="black")+
geom_text(label=paste(round(media,1),
c("c","b","d","a")), vjust=-4)+
ylim(c(0,40))+theme_bw()+theme_classic()+ylab("Resposta")+xlab(" ")+
geom_errorbar(aes(ymax=media+desvio,
ymin=media-desvio), width=0.25)grid.arrange(a,b,c,ncol=3)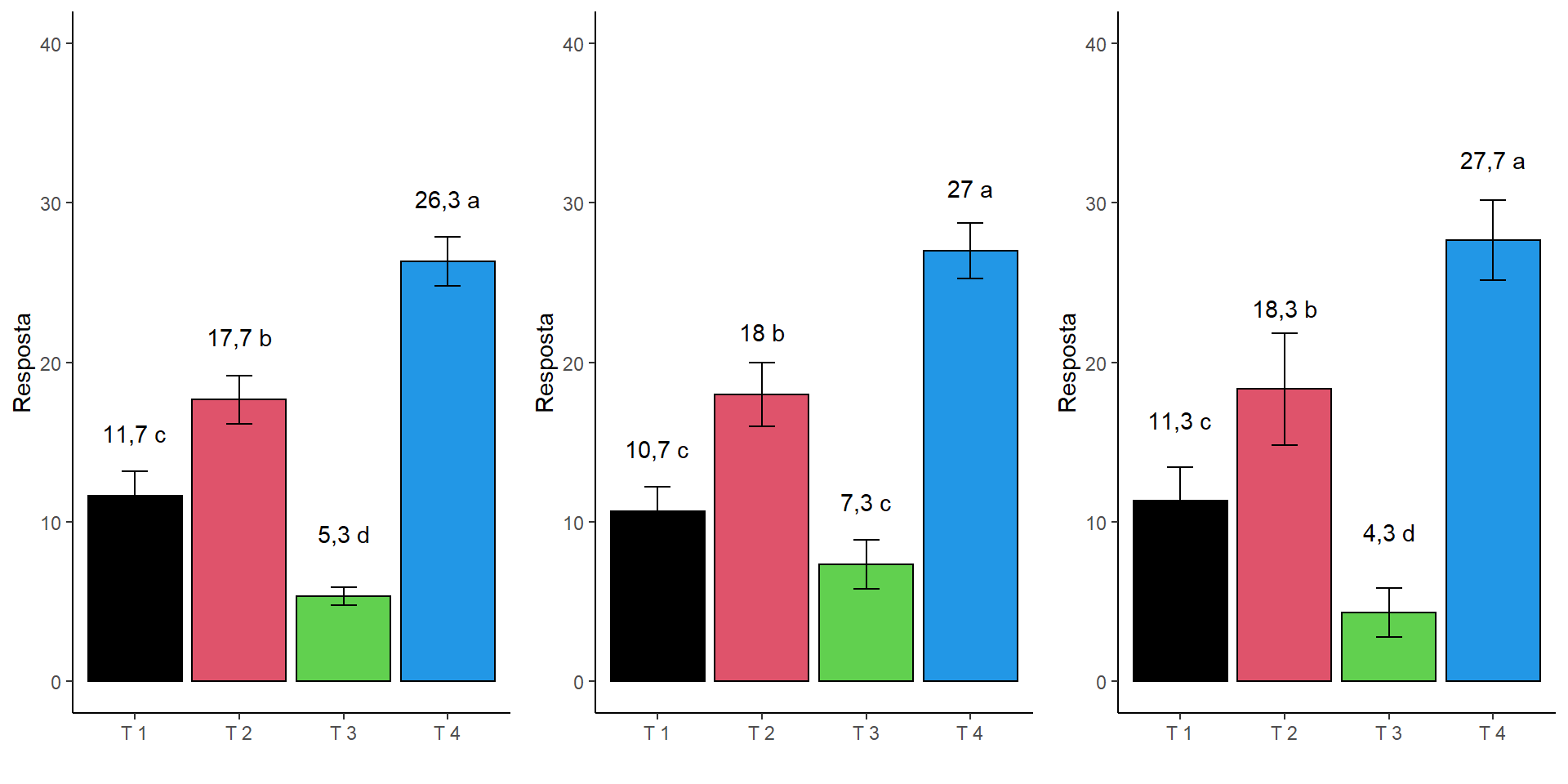
2.10.16 Outra forma de fazer no ggplot2
Conjunto de dados
tratamentos=rep(c(paste("T",1:5)),e=4)
resp=rnorm(20,4,1)
dados=data.frame(tratamentos,resp)Calculando média, desvio-padrão e erro padrão
media=tapply(resp,tratamentos, mean)
desvio=tapply(resp,tratamentos, sd)
erro=desvio/sqrt(4)
data1=data.frame(tratamentos=names(media),
media,
sup=media+erro,
inf=media-erro,
letra=c("a","a","a","a","a"))library(ggplot2)
ggplot(dados,aes(y=resp,x=tratamentos))+
stat_summary(aes(fill=tratamentos),
geom="bar",col="black")+
stat_summary(aes(fill=tratamentos),
geom="errorbar",col="black", width=0.3)+
theme_bw()+
theme(text=element_text(size=14, color="black", family = "serif"))+
ylab("Resposta")+xlab("")+ylim(0,7)+
geom_text(data=data1,aes(y=sup+0.3,
label=paste(format(media,digits=2),letra)))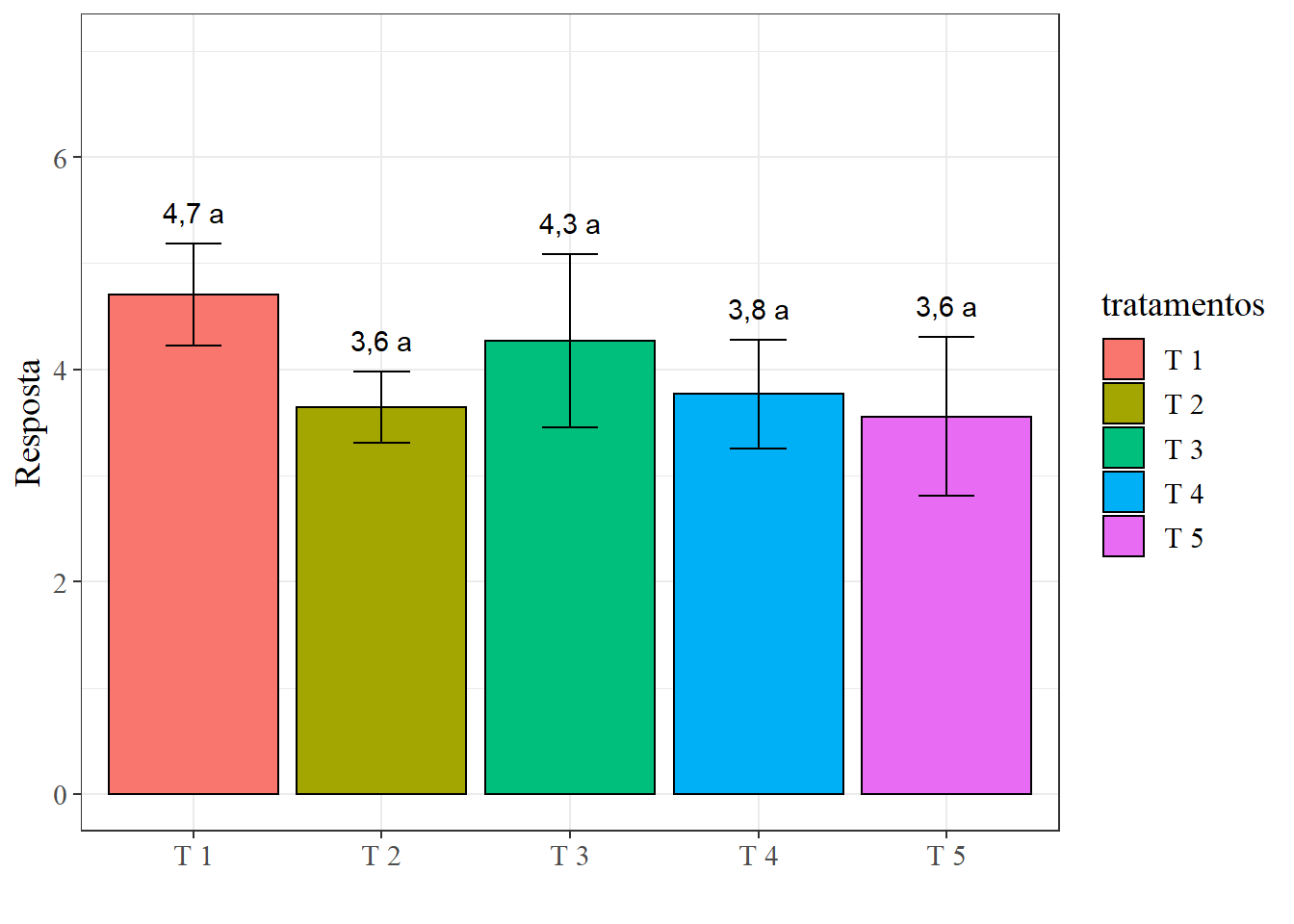
2.11 Pacote ggpubr
Obs. Existem vários packages que utilizam o ggplot2 e geram saídas similares, contudo, com argumentos dos comandos mais simples.
exp1=c(10,12,13,18,19,16,5,6,5,25,26,28)
exp2=c(9,12,11,18,20,16,7,6,9,25,28,28)
exp3=c(9,12,13,18,22,15,3,6,4,25,30,28)
Trat=rep(c(paste("T",1:4)),e=3)
dados=data.frame(Trat,exp1,exp2,exp3)
dados$Trat=as.factor(Trat)library(ggpubr)
library(gridExtra)2.11.1 Comando base
ggbarplot(dados,
x = "Trat",
y = "exp1",
add="mean")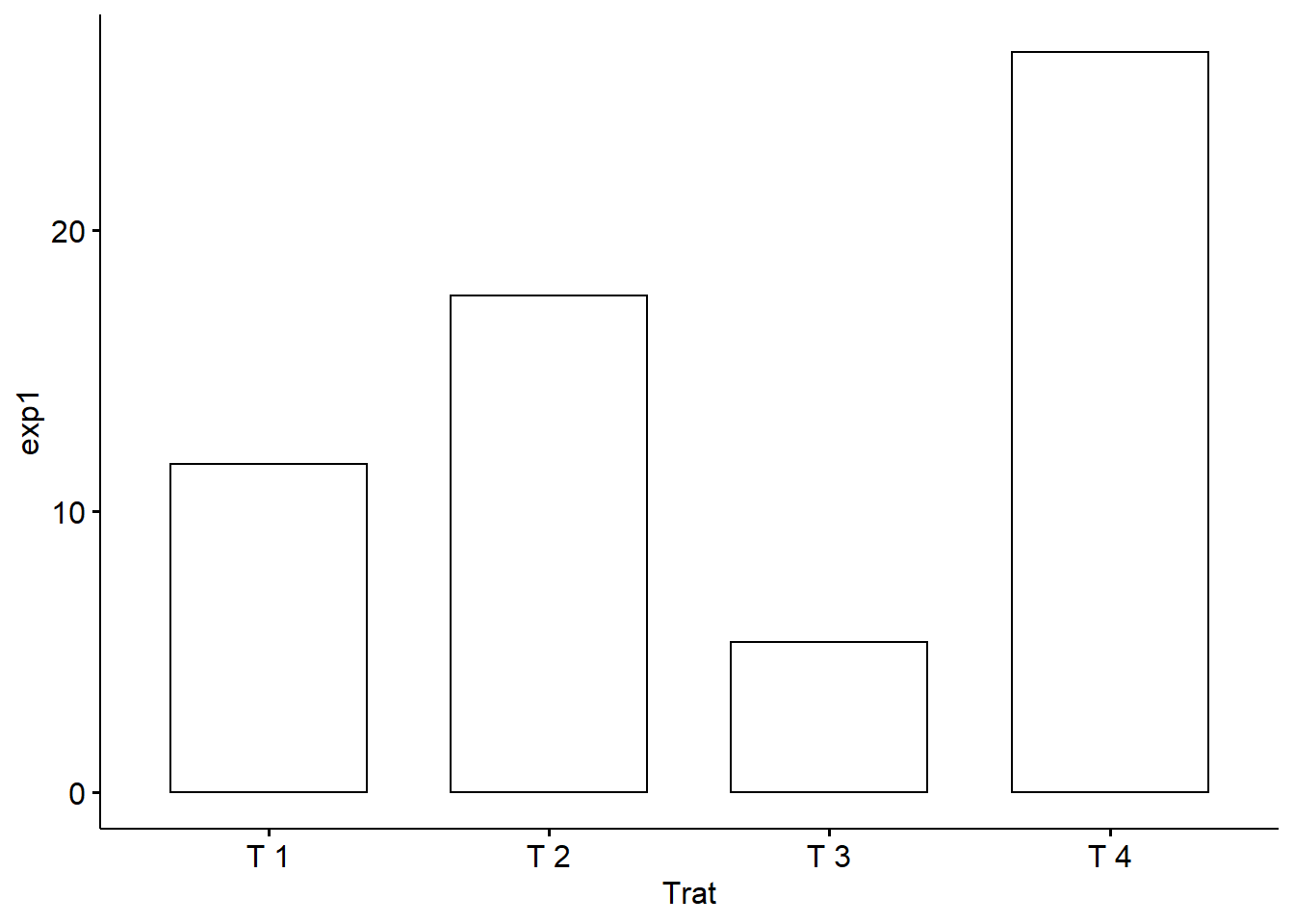
2.11.2 Barras de desvio-padrão
ggbarplot(dados,
x = "Trat",
y = "exp1",
add = "mean_sd")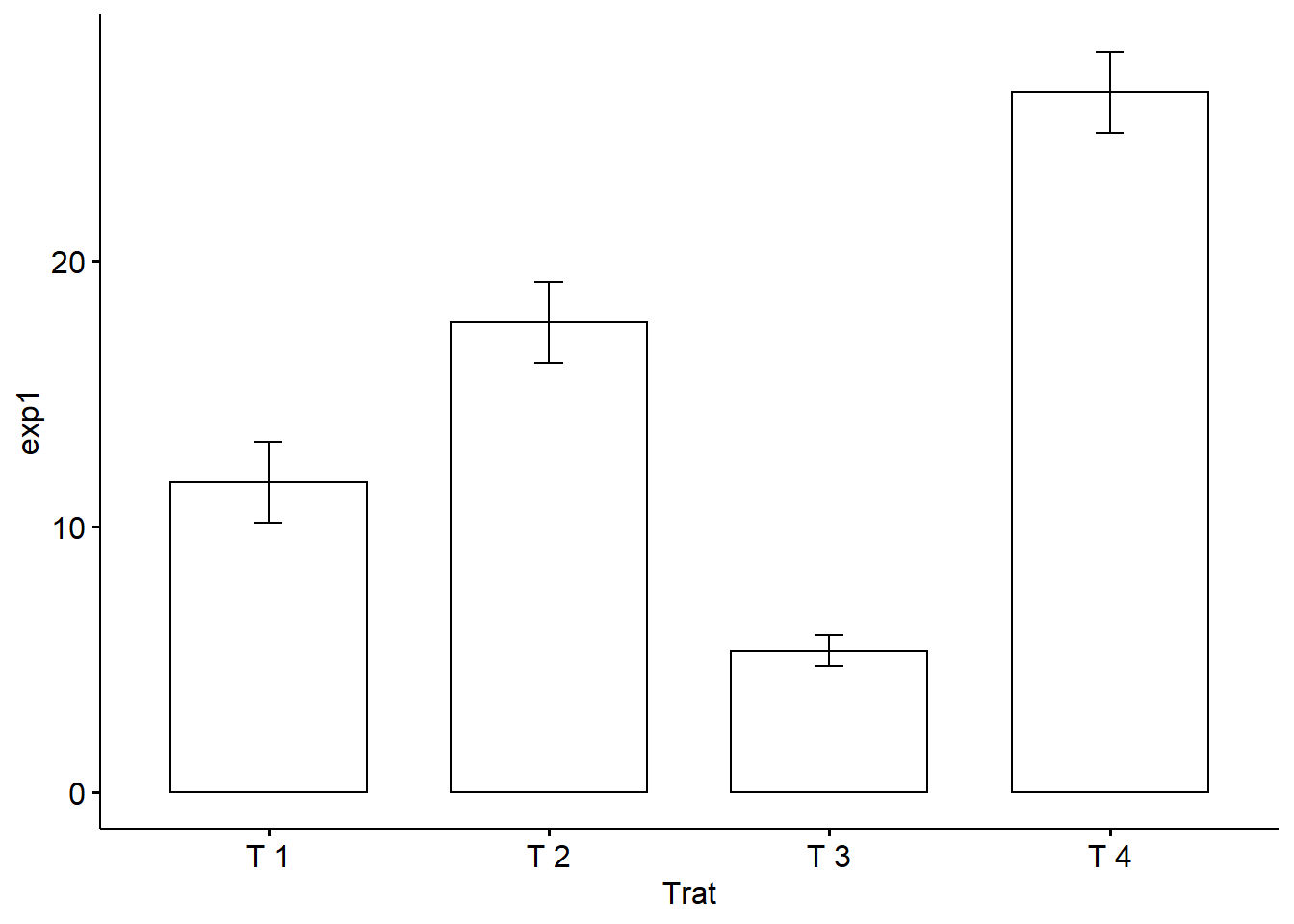
2.11.3 Cor da coluna
ggbarplot(dados,
x = "Trat",
y = "exp1",
add = "mean_sd",
fill = "Trat")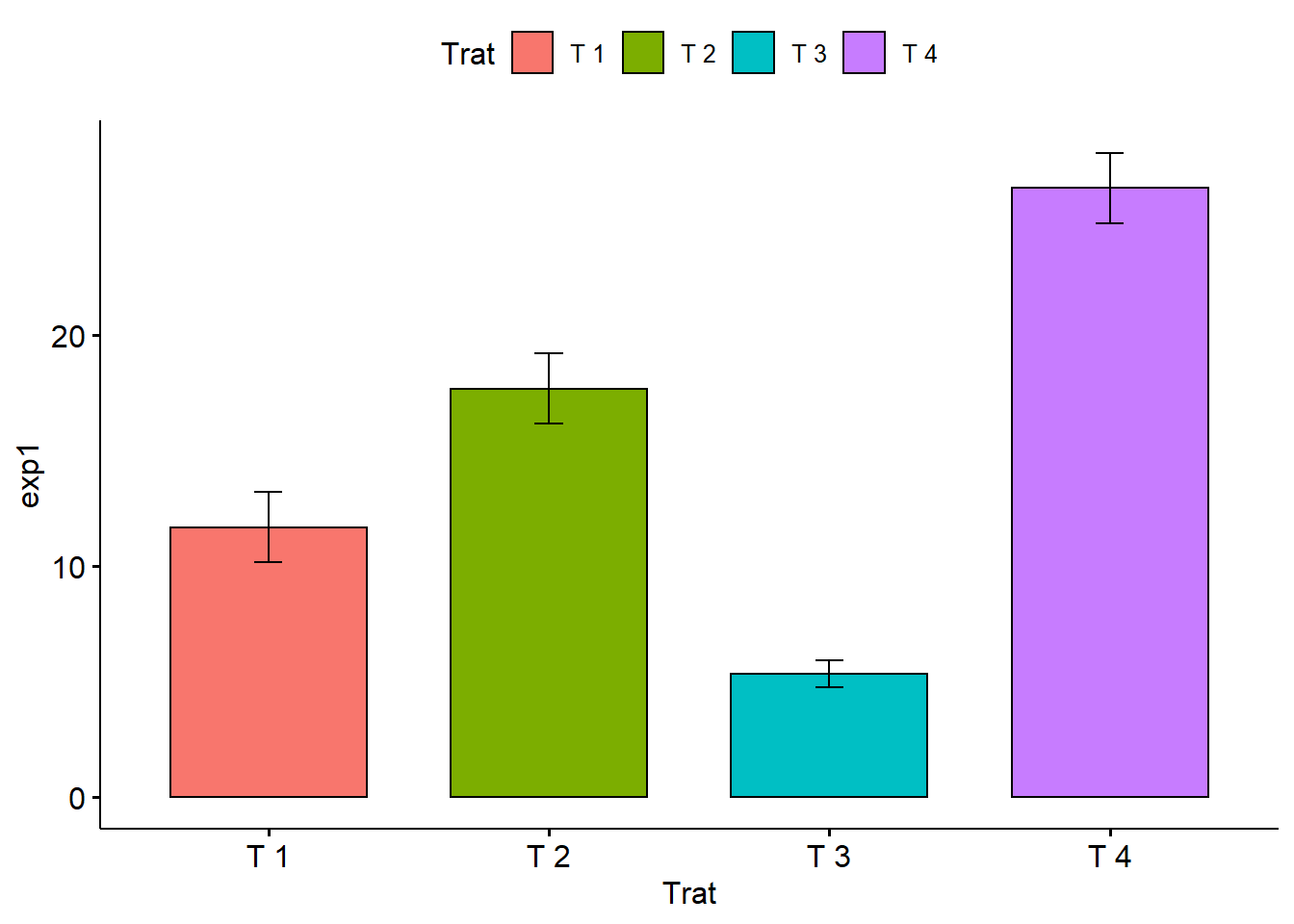
ggbarplot(dados,
x = "Trat",
y = "exp1",
add = "mean_sd",
fill = "Trat",
palette = c(1,2,3,4))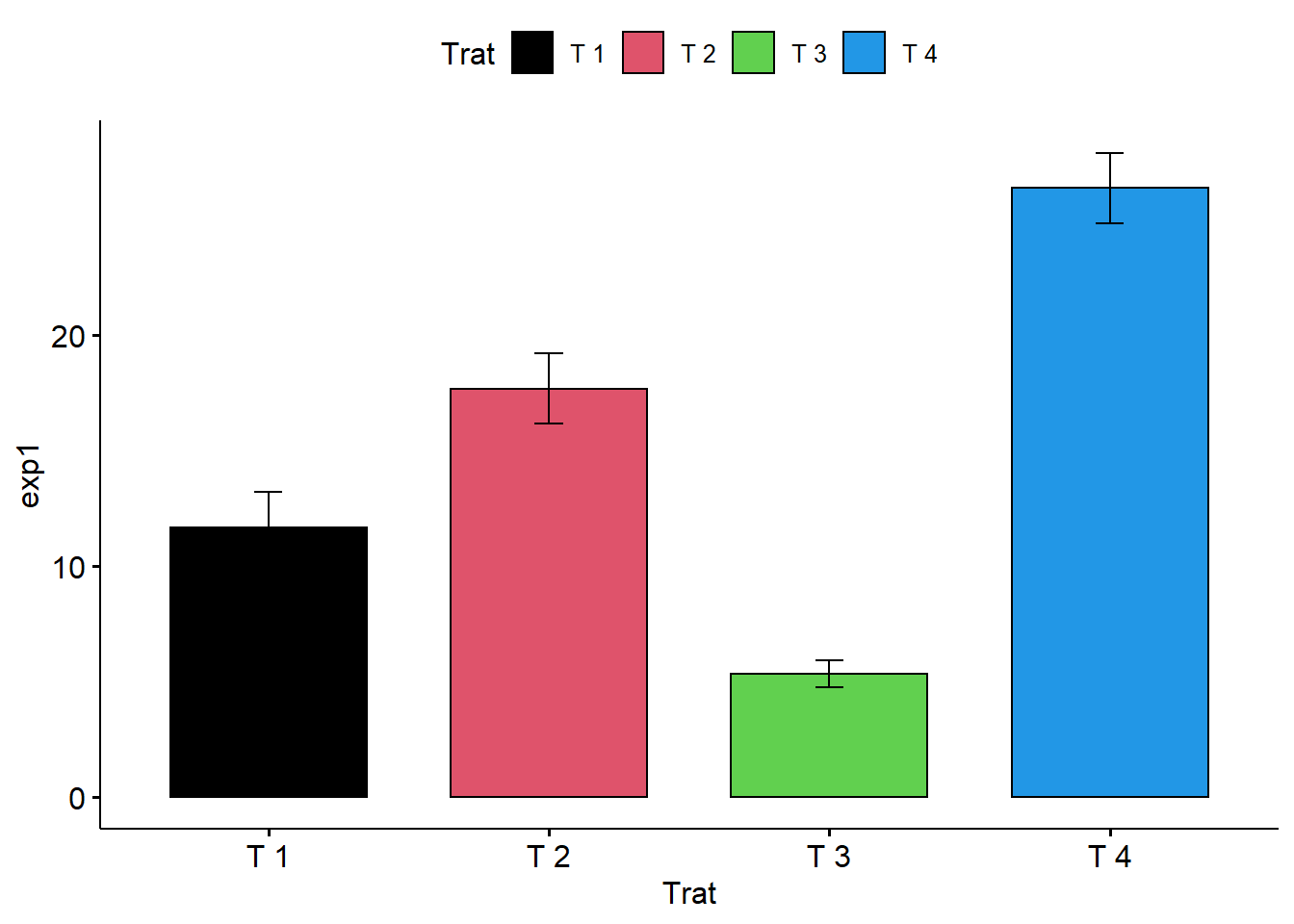
2.11.4 Letra do teste de comparação
ggbarplot(dados,
x = "Trat",
y = "exp1",
add = "mean_sd",
fill = "Trat",
label = c("c","b","d","a"),
lab.vjust=-2)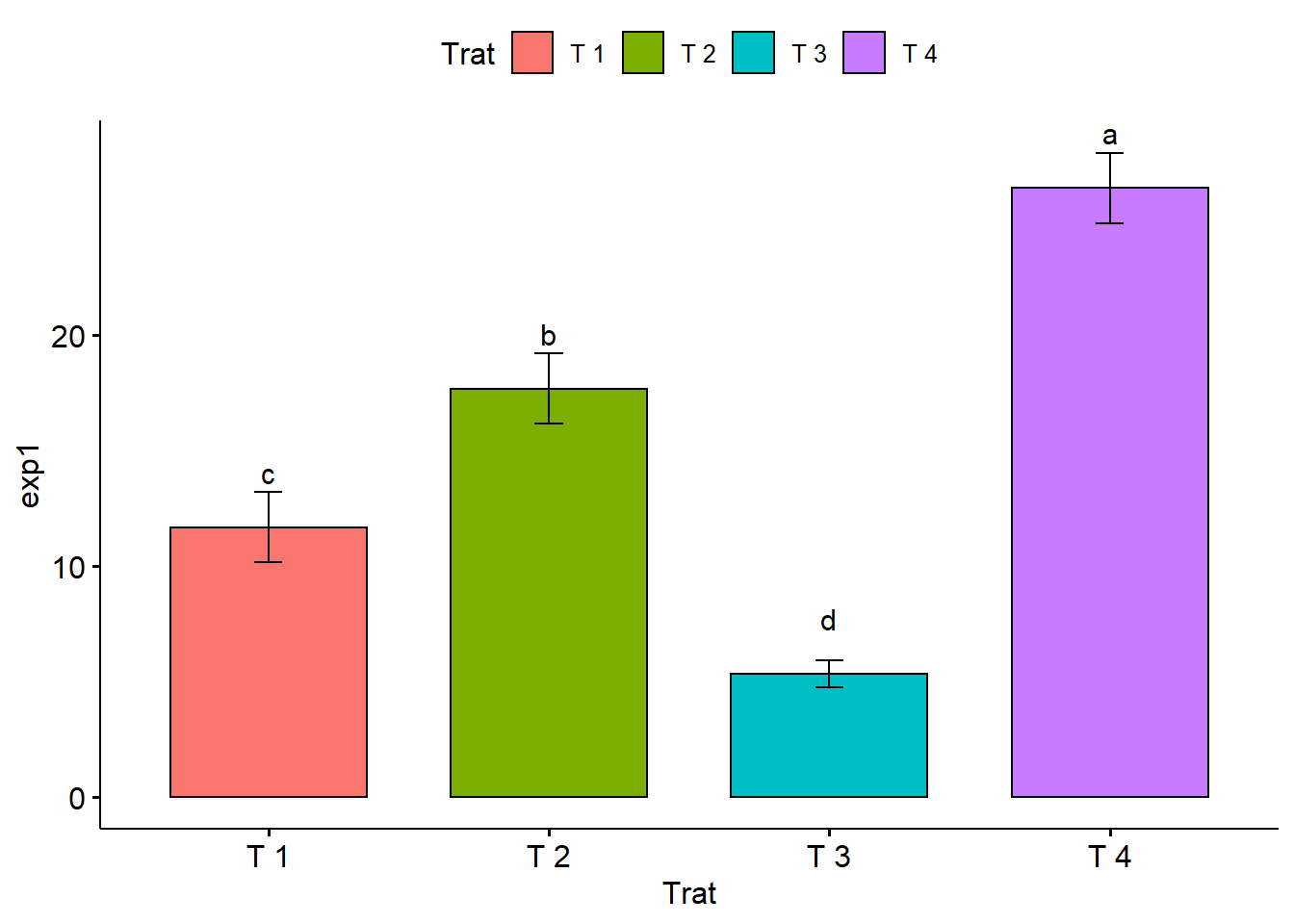
2.11.5 Adicionando a média
media=tapply(exp1,Trat,mean)
ggbarplot(dados,
x = "Trat",
y = "exp1",
add = "mean_sd",
fill = "Trat",
label = paste(round(media,1),c("c","b","c","a")),
lab.vjust=-2)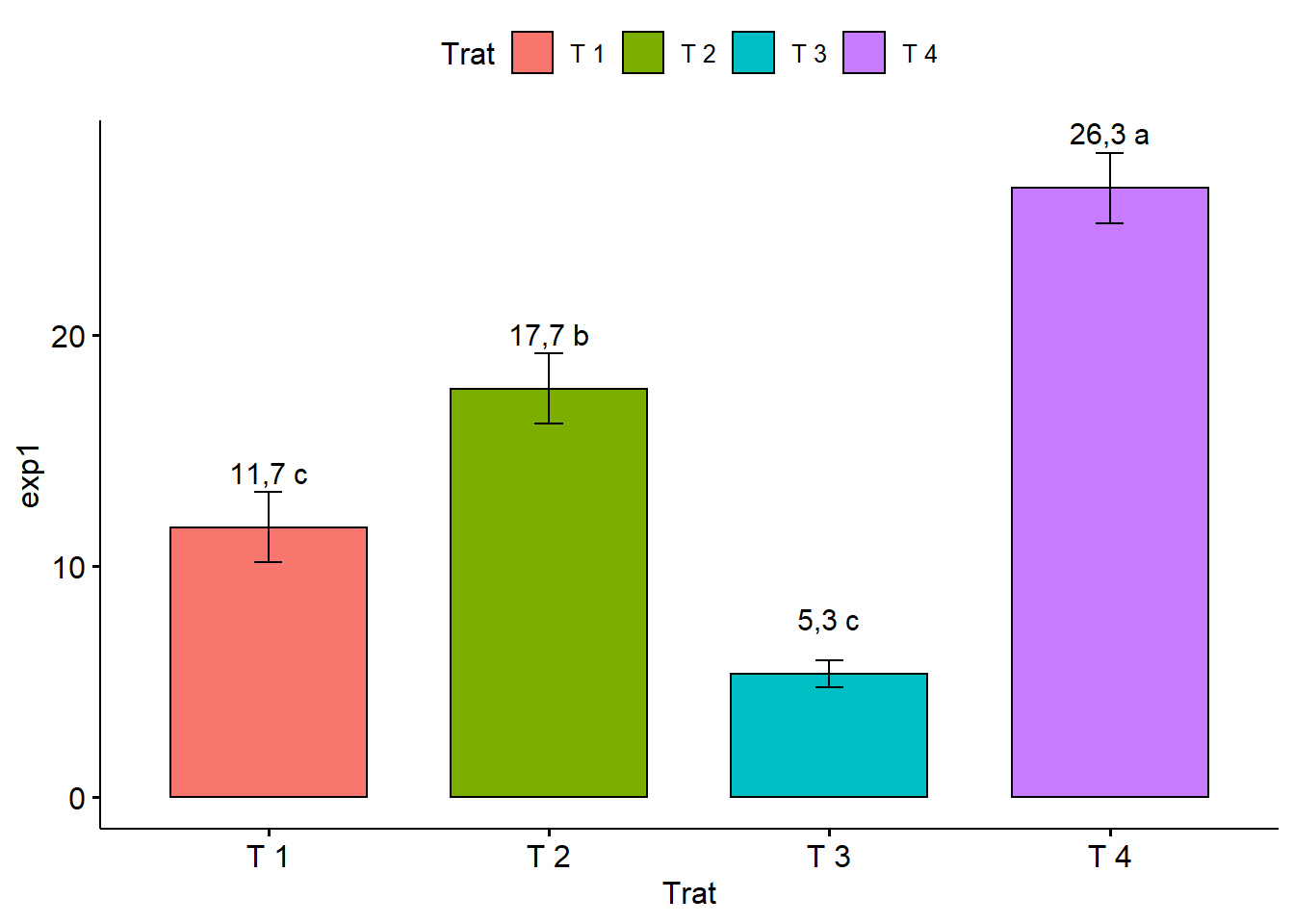
2.11.6 Escala do eixo Y
ggbarplot(dados,
x = "Trat",
y = "exp1",
add = "mean_sd",
fill = "Trat",
label = paste(round(media,1),c("c","b","d","a")),
lab.vjust=-2)+ylim(c(0,40))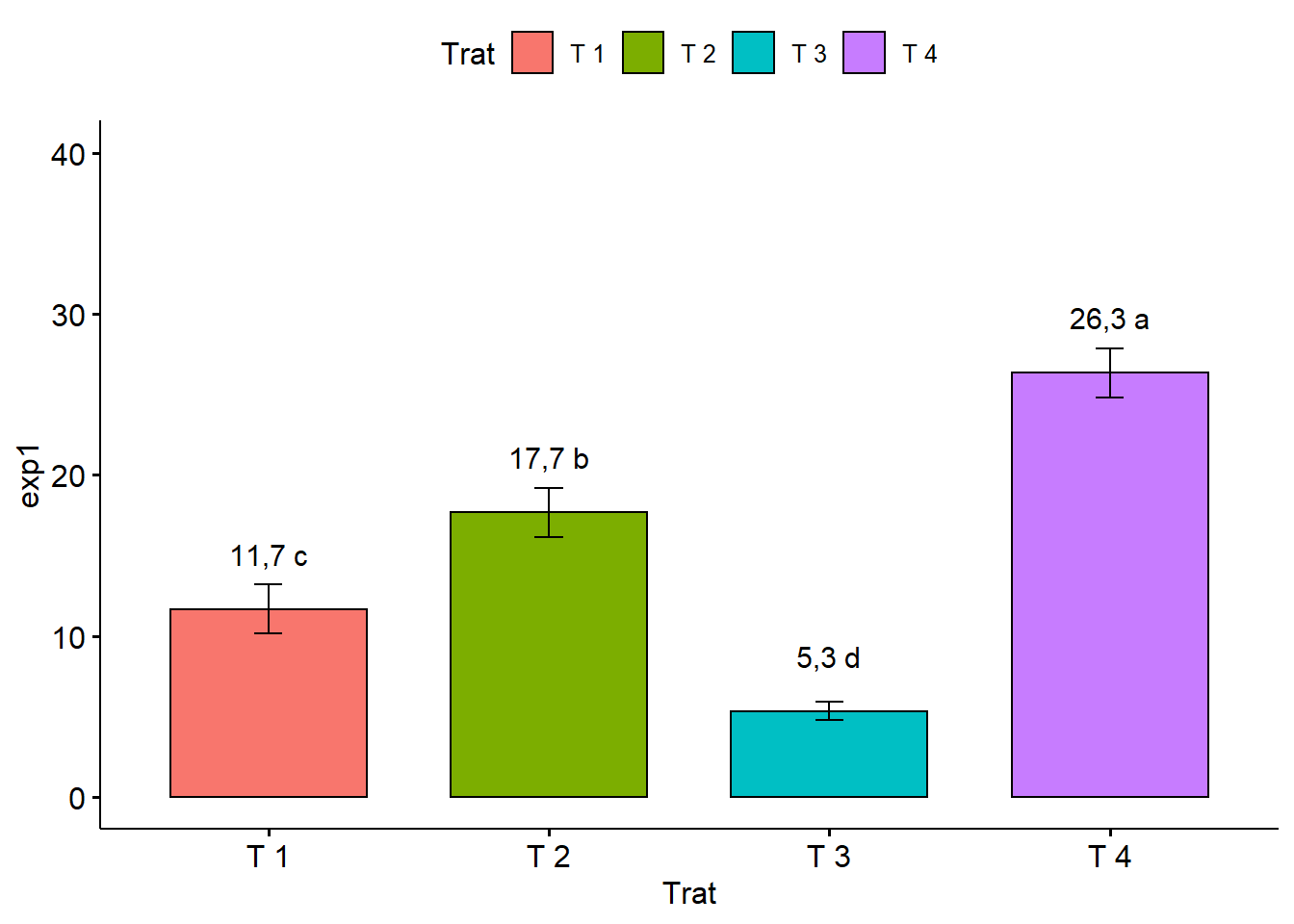
2.11.7 Removendo legenda
ggbarplot(dados,
x = "Trat",
y = "exp1",
add = "mean_sd",
fill = "Trat",
label = paste(round(media,1),c("c","b","d","a")),
lab.vjust=-2,
legend="n")+ylim(c(0,40))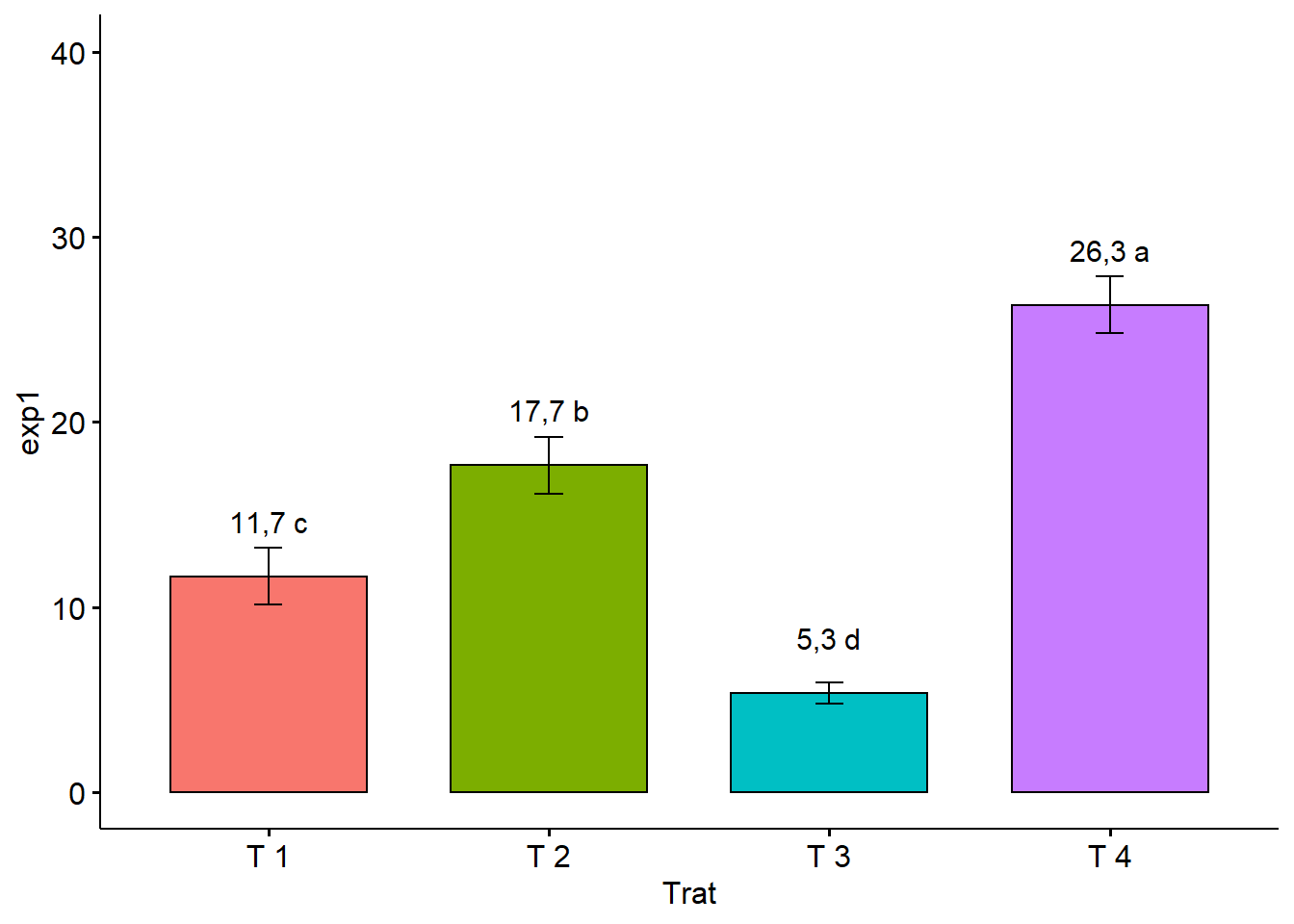
2.11.8 Juntando os gráficos
2.11.9 Variável exp1
media=tapply(exp1,Trat,mean)
a=ggbarplot(dados,
x = "Trat",
y = "exp1",
add = "mean_sd",
fill = "Trat",
label = paste(round(media,1),c("c","b","d","a")),
lab.vjust=-3,
legend="n")+ylim(c(0,40))2.11.10 Variável exp2
media=tapply(exp2,Trat,mean)
b=ggbarplot(dados,
x = "Trat",
y = "exp2",
add = "mean_sd",
fill = "Trat",
label = paste(round(media,1),c("c","b","d","a")),
lab.vjust=-3,
legend="n")+ylim(c(0,40))2.11.11 Variável exp3
media=tapply(exp3,Trat,mean)
c=ggbarplot(dados,
x = "Trat",
y = "exp3",
add = "mean_sd",
fill = "Trat",
label = paste(round(media,1),c("c","b","d","a")),
lab.vjust=-3,
legend="n")+ylim(c(0,40))grid.arrange(a,b,c,ncol=3)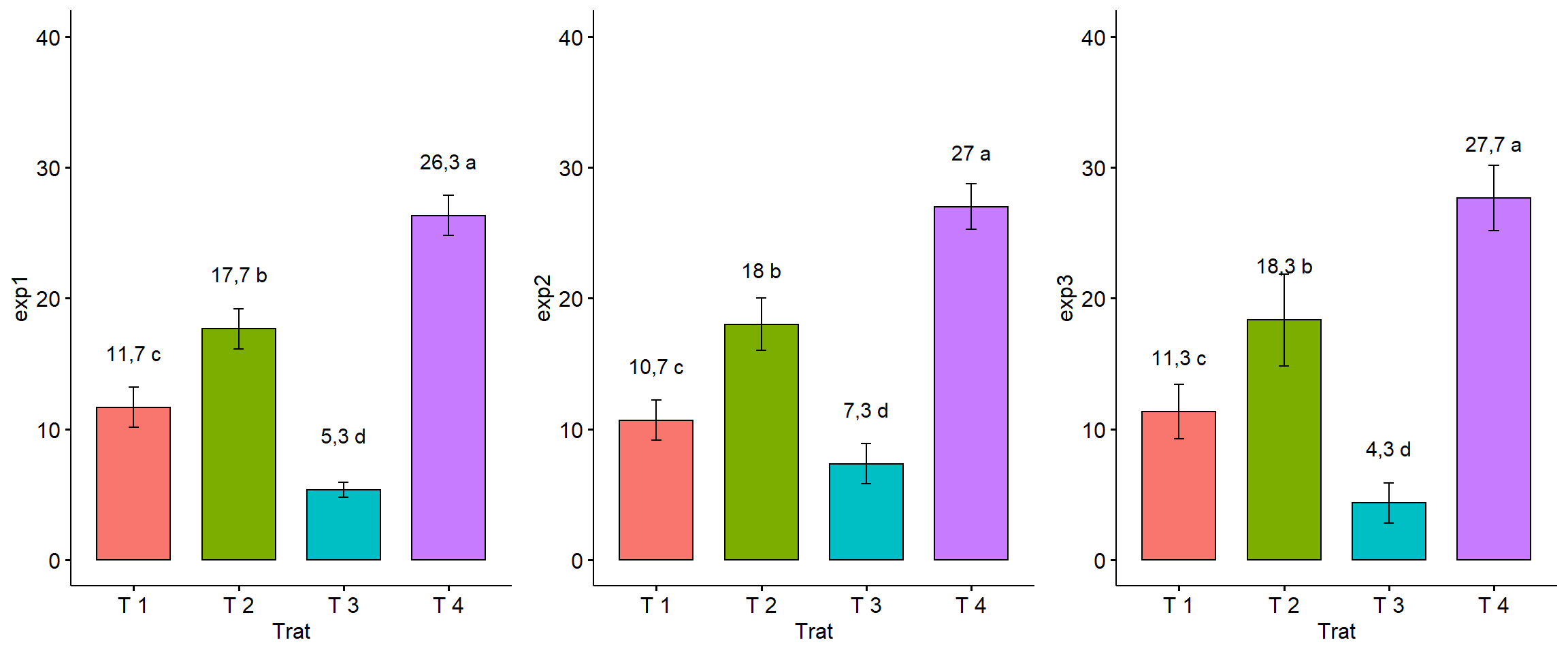
Como deixar apenas o gráfico a esquerda com a escala de Y?
Existem casos em que uma mesma variável foi analisada em várias situações e dessa forma, geramos gráficos com a mesma unidade de medida. Nesse sentido, é frequente apresentar apenas uma escala de Y, geralmente o gráfico a esquerda. No pacote ggpubr, podemos efetuar da seguinte forma:
media=tapply(exp1,Trat,mean)
a=ggbarplot(dados,
x = "Trat",
y = "exp1",
add = "mean_sd",
fill = "Trat",
ylab="Resposta",
label = paste(round(media,1),c("c","b","d","a")),
lab.vjust=-3,
legend="n")+ylim(c(0,40))media=tapply(exp2,Trat,mean)
b=ggbarplot(dados,
x = "Trat",
y = "exp2",
add = "mean_sd",
fill = "Trat",
label = paste(round(media,1),c("c","b","c","a")),
lab.vjust=-3,
legend="n",
yscale="n")+
ylim(c(0,40))+
theme(axis.text.y=element_blank())+ # Comando para remover os números da escala de Y
ylab("") # Remover nome do eixo Y media=tapply(exp3,Trat,mean)
c=ggbarplot(dados,
x = "Trat",
y = "exp3",
add = "mean_sd",
fill = "Trat",
label = paste(round(media,1),c("c","b","d","a")),
lab.vjust=-4,
legend="n")+
ylim(c(0,40))+
theme(axis.text.y=element_blank())+
ylab("")grid.arrange(a,b,c,ncol=3)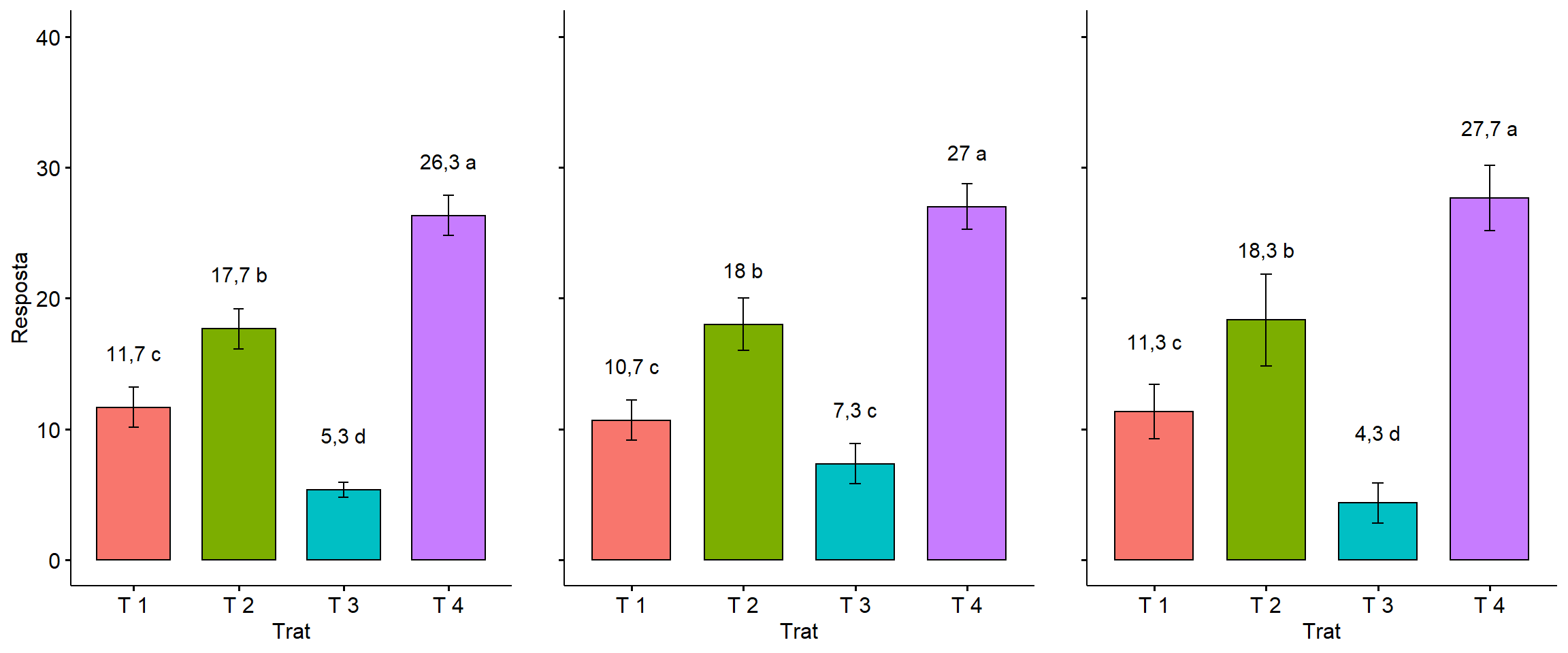
2.12 Duas variáveis categóricas
2.12.1 Conjunto de dados
Fator1=factor(rep(c(paste("F",1:2)),e=20))
Fator2=factor(c(rep(c(paste("T",1:5)),e=4),rep(c(paste("T",1:5)),e=4)))
resposta=c(100,120,110,90,150,145,149,165,250,244,220,239,220,206,210,210,266,249,248,260,110,130,120,100,160,165,169,175,160,154,144,149,230,216,220,220,276,259,258,270)
dados=data.frame(Fator1,Fator2,resposta)
## Média e Desvio-padrão (Por Tratamento)
media=with(dados, tapply(dados$resposta,list(Fator1, Fator2), mean))
desvio=with(dados, tapply(resposta,list(Fator1, Fator2), sd))2.12.2 Gráfico simples
barplot(media, beside = T)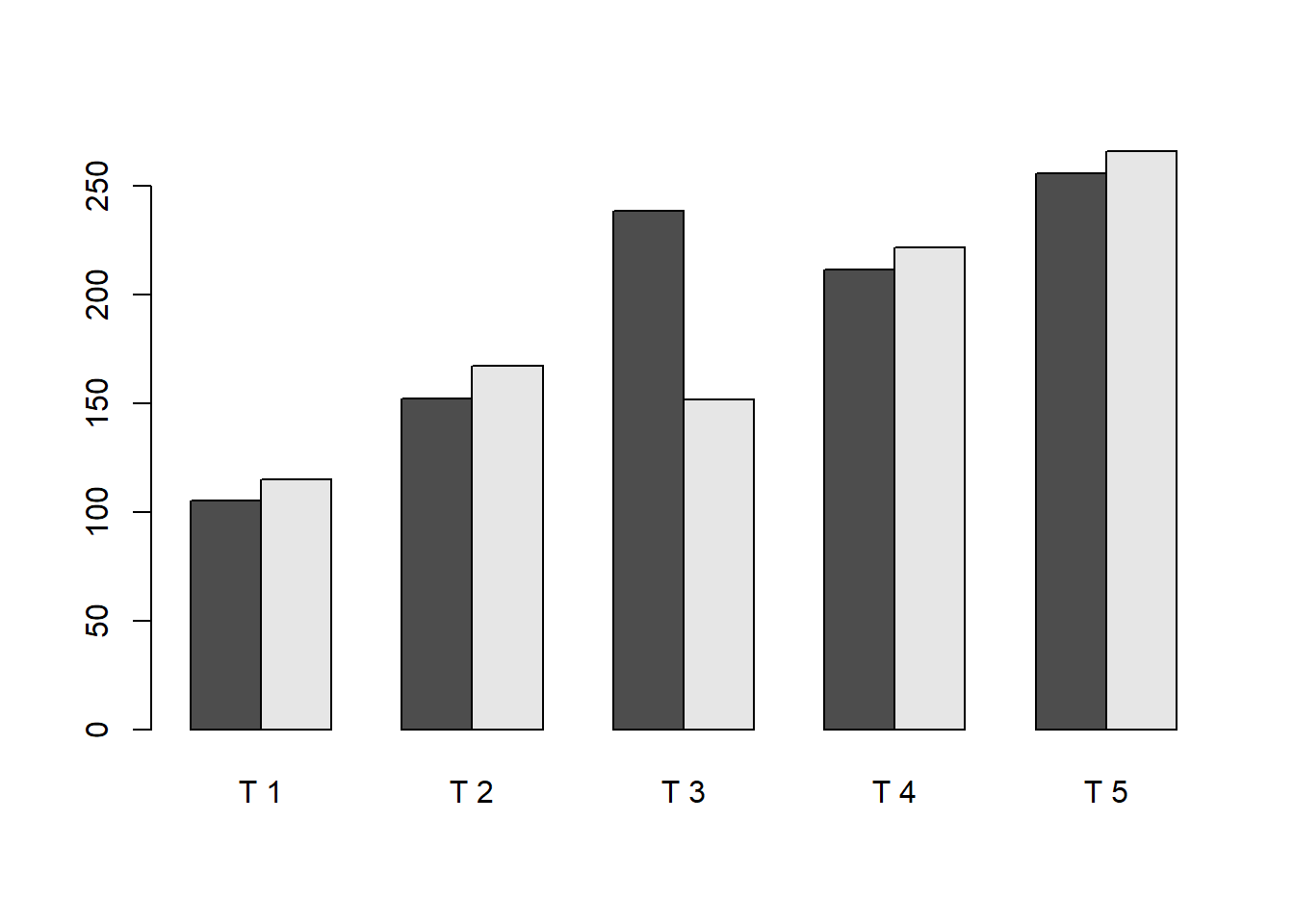
O argumento beside=T é refente a um gráfico de barras em que as barras são posicionadas lado a lado. Do contrário, as barras serão empilhadas (stacked).
2.12.3 Melhorias
barplot(media,
beside = T,
las=1,
col=c("lawngreen","gold"),
ylab="Resposta",
xlab="Fator2",
ylim=c(0,300))
abline(h=0)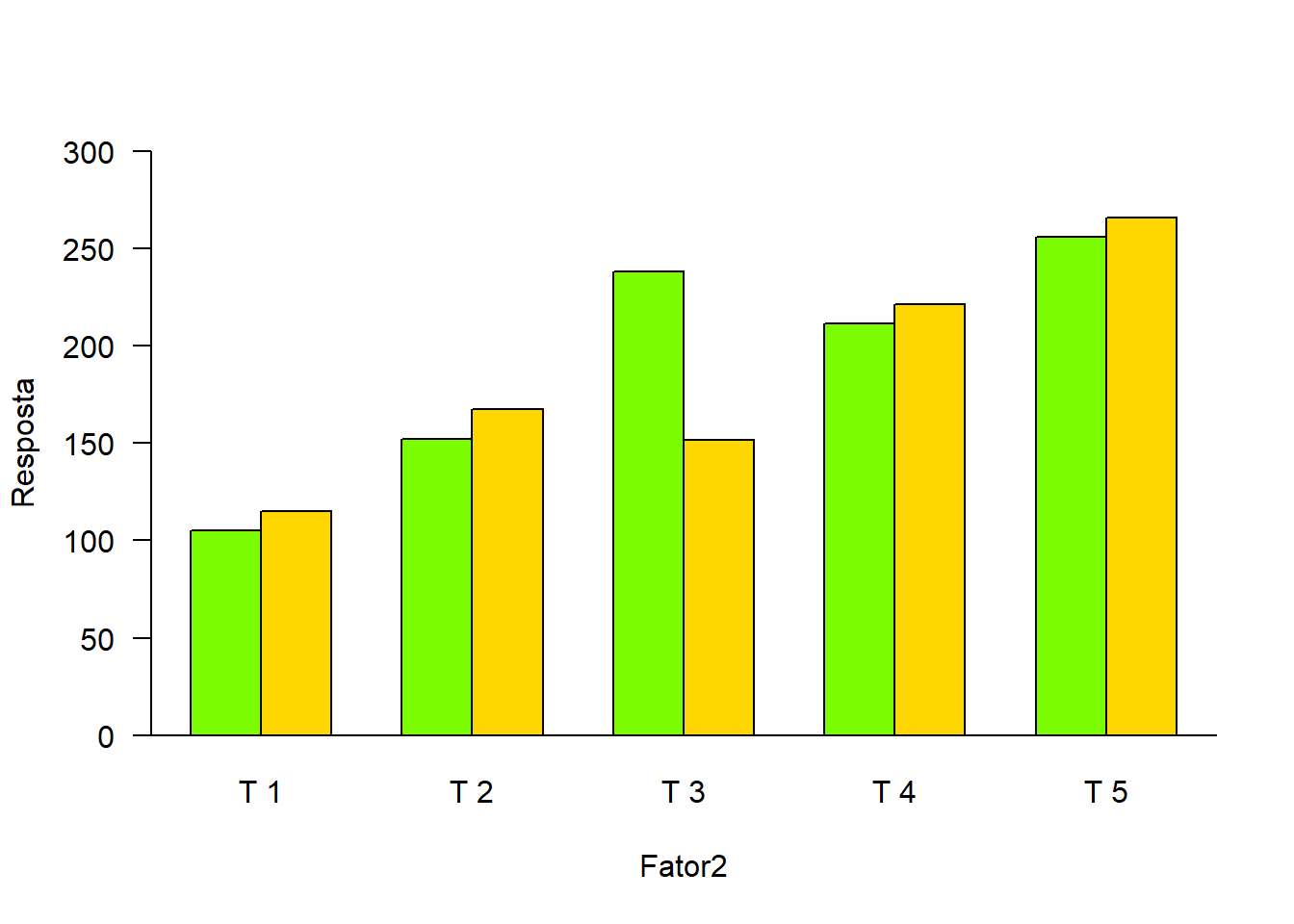
Comandos:
las=1: deixar escala do eixo Y na vertical
col=“cor”: mudar cor das barras (Ex. “red,”“blue,”“green” ou gray.colors(quantidade de tonalidades) para escala cinza ou rainbow(quantidade de cores) para escala colorida. Também é possível específicar a cor de cada barra (col=c(“red,”“green,”“yellow,”“gray,”“blue”))).
xlab e ylab: nomear eixo X e Y
xlim e ylim: escala do eixo X e Y
abline(h=0): linha na horizontal em Y=0 (No caso de vertical, abline(v=0)). É possível alterar a cor pela função “col=”cor"" e o tracejado pelo “lty=número” (Ver o Help do comando)
2.12.4 Barras de desvio-padrão
bar=barplot(media,
beside=T,
las=1,
ylab="Resposta",
xlab="Tratamentos",
ylim=c(0,300))
abline(h=0)
arrows(bar,media+desvio,bar,media-desvio,length = 0.1,angle=90,code=3)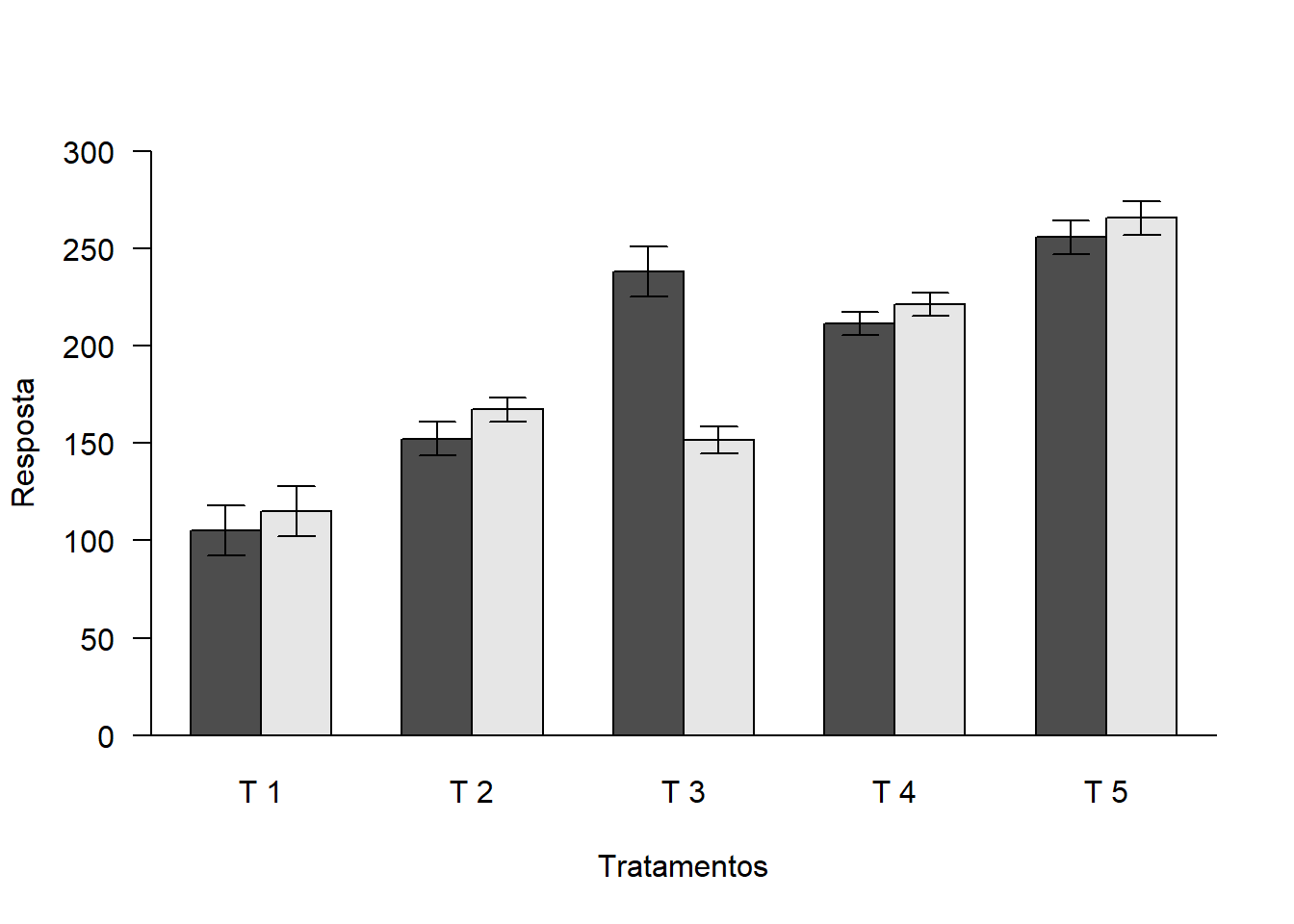
2.12.5 Unidade do eixo Y
(Ex. \(Kg\ ha^{-1}\))
bar=barplot(media, beside=T,
las=1,
ylab=expression("Resposta"~(kg~ha^-1)),
xlab="Tratamentos",
ylim=c(0,300))
abline(h=0)
arrows(bar,media+desvio,bar,media-desvio,length = 0.1,angle=90,code=3)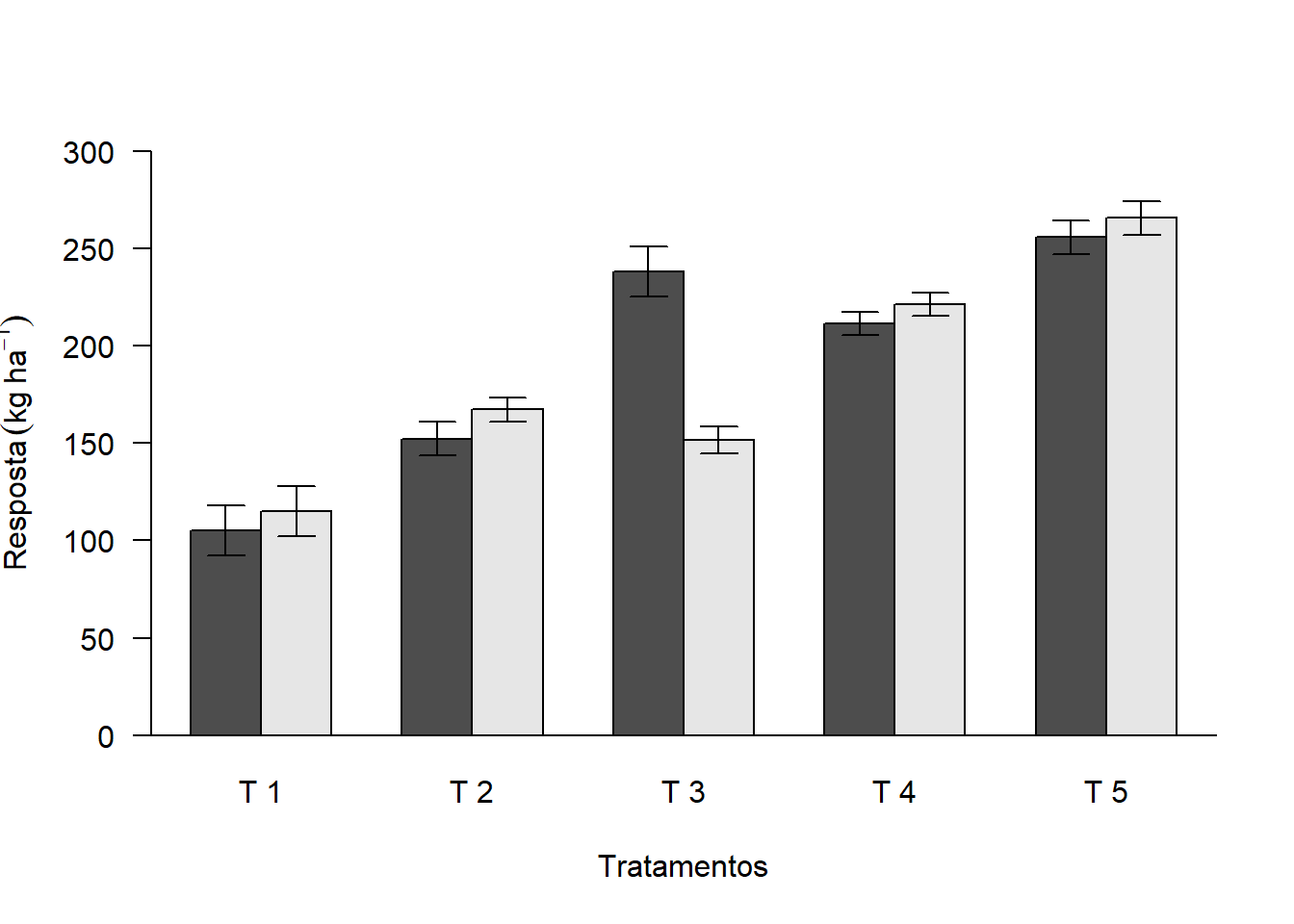
2.12.6 Média acima das barras
bar=barplot(media,
beside=T,
las=1,
ylab=expression("Resposta"~(kg~ha^-1)),
xlab="Tratamentos",
ylim=c(0,300))
abline(h=0)
text(bar,media+desvio+10,media, cex=0.8)
arrows(bar,media+desvio,bar,media-desvio,length = 0.1,angle=90,code=3)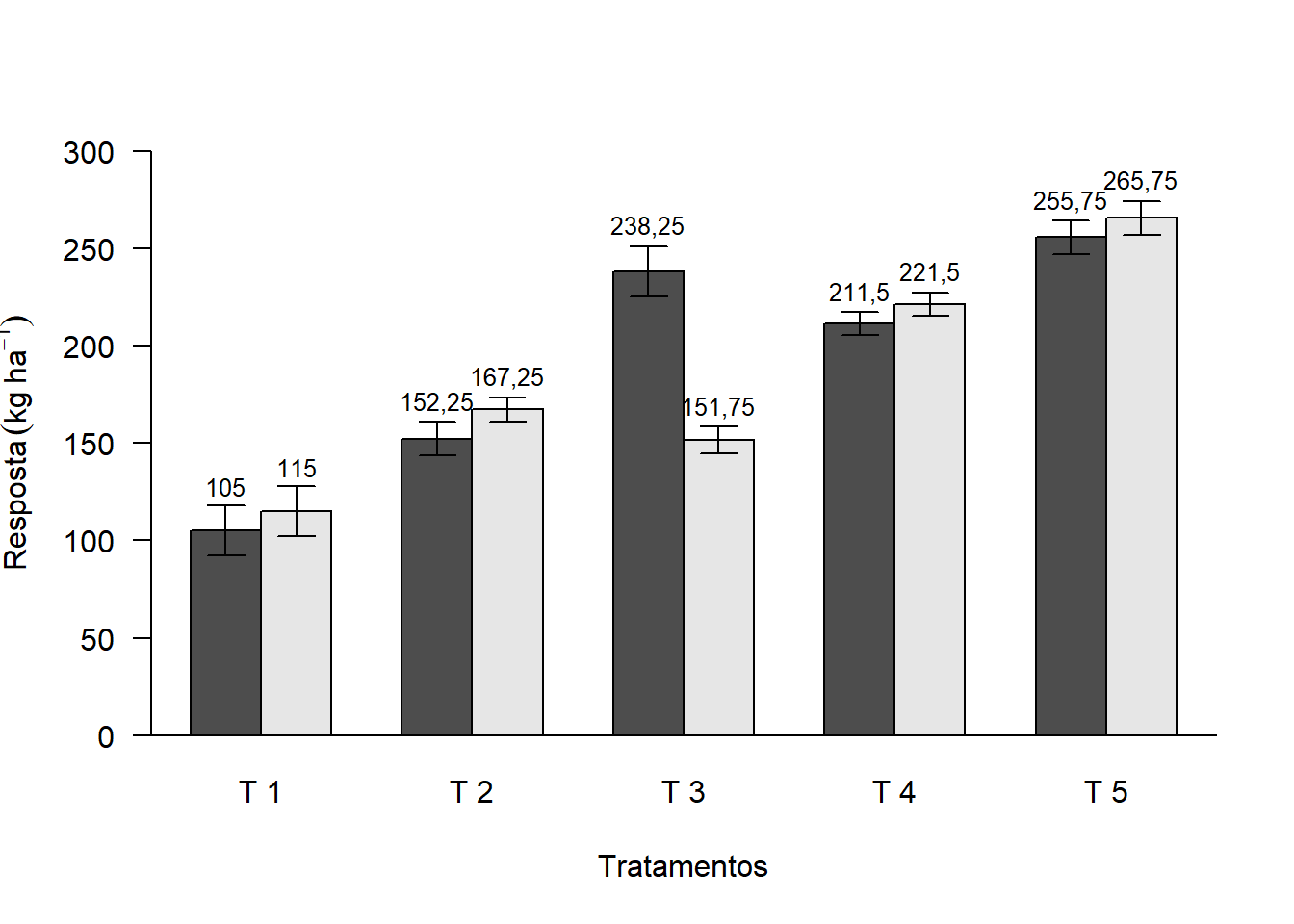
2.12.7 Separação de casa decimal
options(OutDec=",")
bar=barplot(media, beside=T,
las=1,
ylab=expression("Resposta"~(kg~ha^-1)),
xlab="Tratamentos",
ylim=c(0,300))
abline(h=0)
text(bar,media+desvio+10,media, cex=0.8)
arrows(bar,media+desvio,bar,media-desvio,length = 0.1,angle=90,code=3)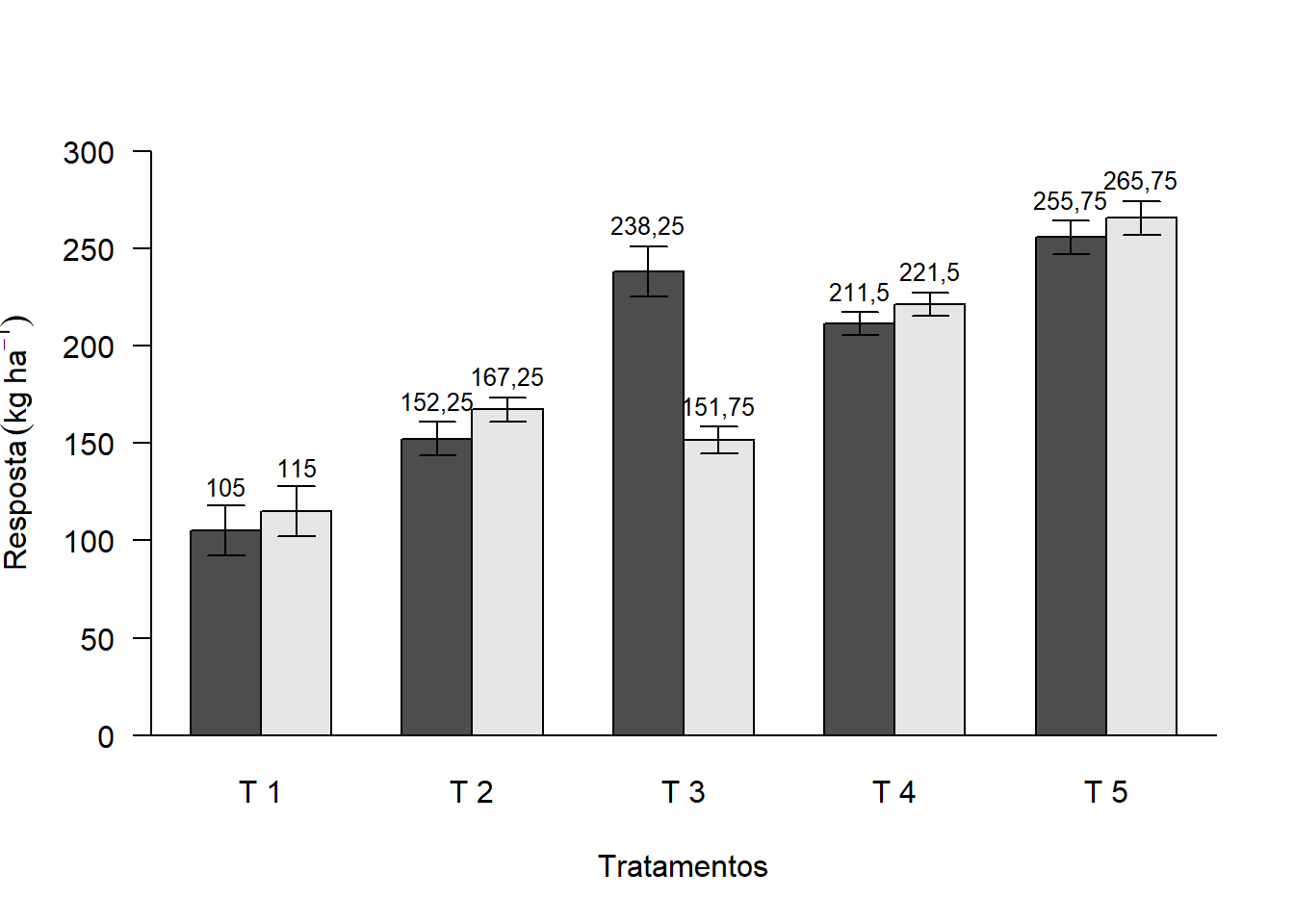
2.12.8 Letras do teste de comparação
tukey=c("dB","dA","cB","cA","cB","cA","bB","bA","aB","aA")
options(OutDec=",")
bar=barplot(media, beside=T,
las=1,
ylab=expression("Resposta"~(kg~ha^-1)),
xlab="Tratamentos",
ylim=c(0,300))
abline(h=0)
text(bar,media+desvio+10,paste(round(media,0),tukey), cex=0.8)
arrows(bar,media+desvio,bar,media-desvio,length = 0.1,angle=90,code=3)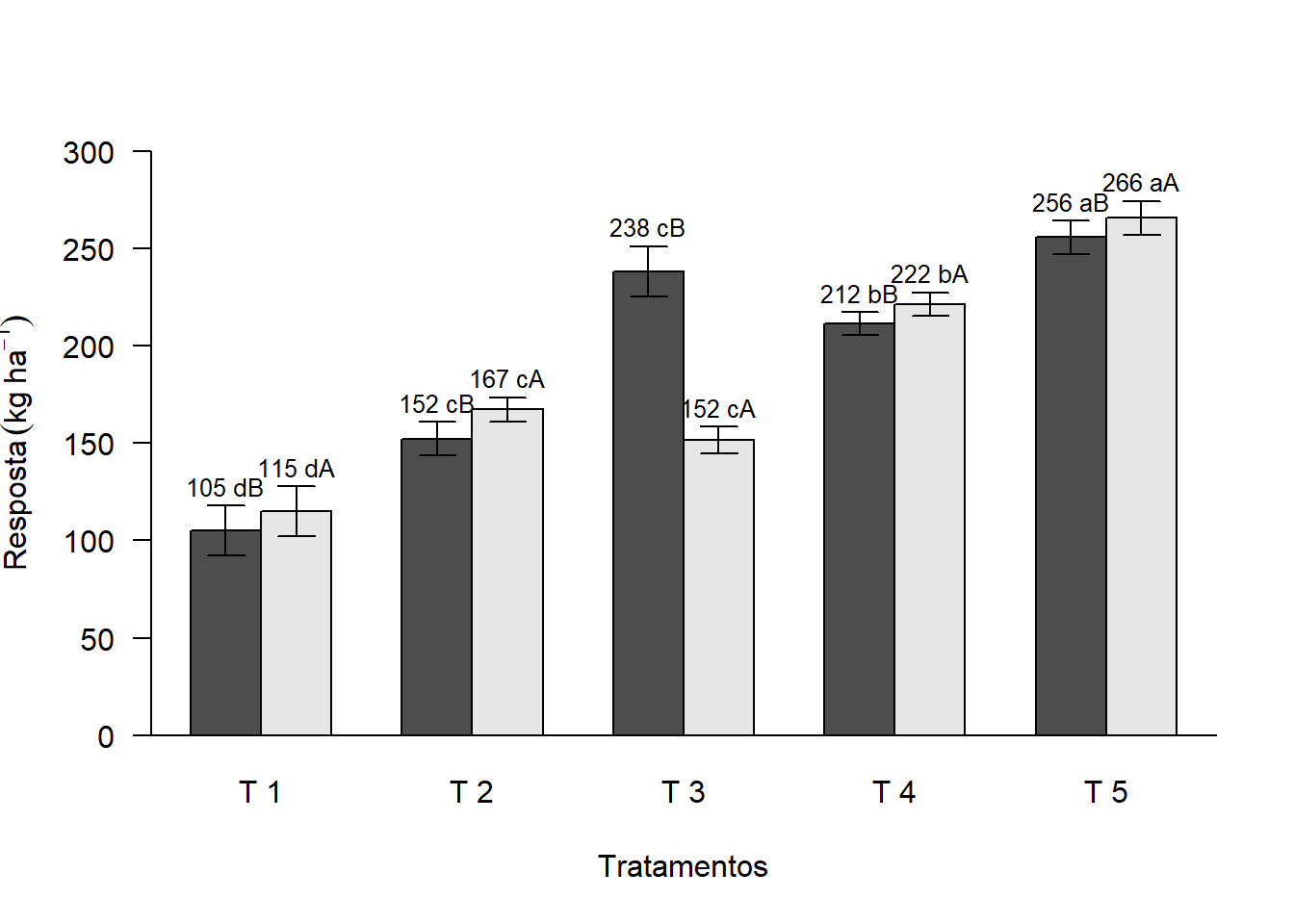
2.12.9 Adicionando legenda
legend.text=rownames(media): adicionar a legenda (neste caso em relação ao Fator 2)
args.legend: argumentos da legenda (x=“topleft”: legenda será adicionada no parte superior esquerda, podemos adicionar superior direito (“topright”), inferior esquerdo (“bottomleft”), inferior direito (“bottomright”), centralizado (“center”))
tukey=c("dB","dA","cB","cA","cB","cA","bB","bA","aB","aA")
options(OutDec=",")
bar=barplot(media,
beside=T,
legend.text = rownames(media),
args.legend = list(x="topleft", bty="n"),
las=1,
ylab=expression("Resposta"~(kg~ha^-1)),
xlab="Tratamentos",
ylim=c(0,300))
abline(h=0)
text(bar,media+desvio+10,paste(round(media,0),tukey), cex=0.8)
arrows(bar,media+desvio,bar,media-desvio,length = 0.1,angle=90,code=3)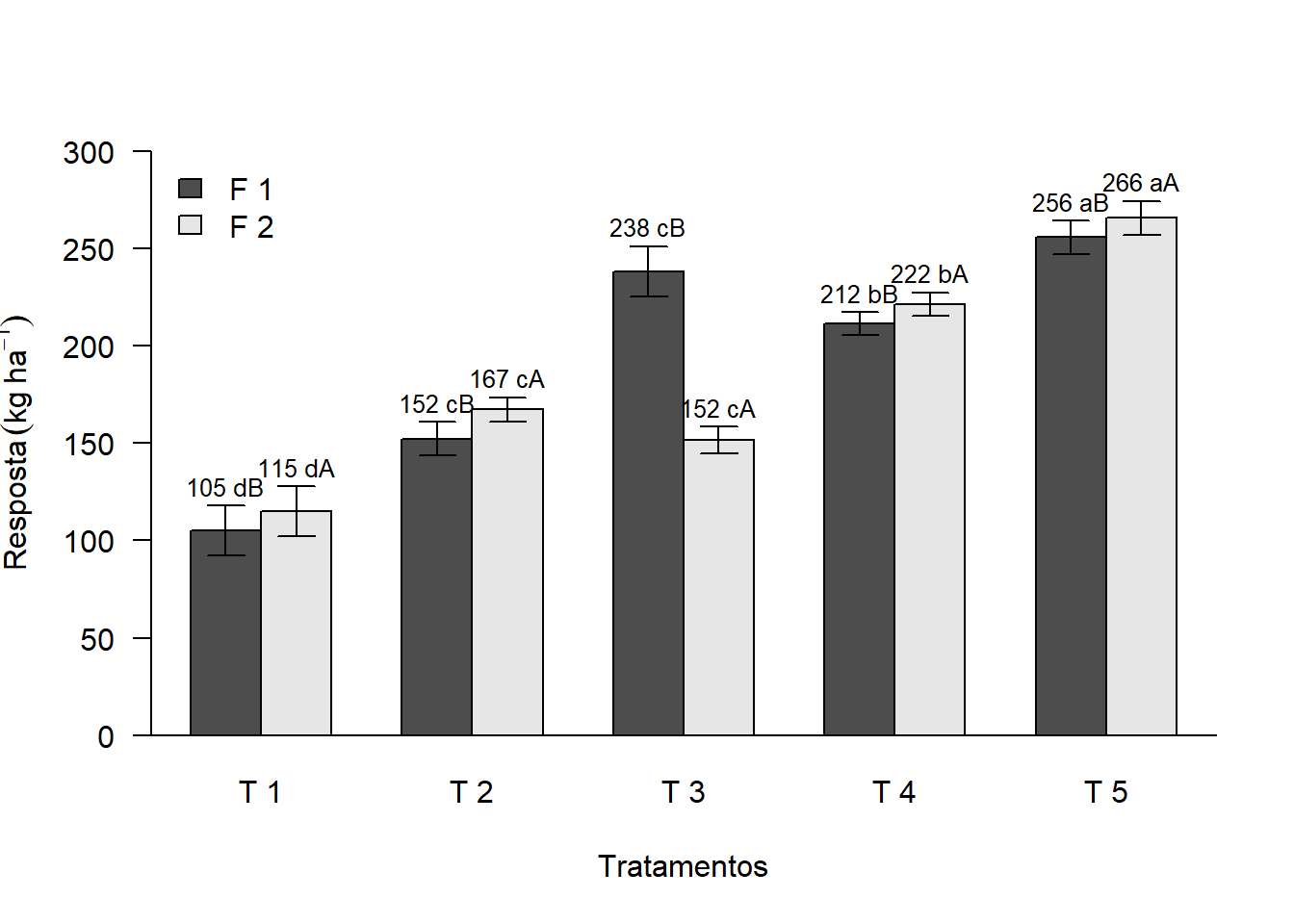
2.13 Colunas empilhadas
2.13.1 Conjunto de dados
Fator1=factor(rep(c(paste("F",1:2)),e=20))
Fator2=factor(c(rep(c(paste("T",1:5)),e=4),rep(c(paste("T",1:5)),e=4)))
resposta=c(100,120,110,90,150,145,149,165,250,244,220,239,220,206,210,
210,266,249,248,260,110,130,120,100,160,165,169,175,160,154,
144,149,230,216,220,220,276,259,258,270)
dados=data.frame(Fator1,Fator2,resposta)
## Média e Desvio-padrão (Por Tratamento)
media=with(dados, tapply(dados$resposta,list(Fator1, Fator2), mean))
desvio=with(dados, tapply(resposta,list(Fator1, Fator2), sd))2.13.2 Gráfico básico
barplot(media, beside=F)
O argumento beside=F é refente a um gráfico de barras em que as barras são posicionadas lado a lado. Do contrário, as barras serão empilhadas (stacked).
2.13.3 Melhorias
barplot(media,
beside=F,
las=1,
col=c("lawngreen","gold"),
ylab="Resposta",
xlab="Fator2",
ylim=c(0,600))
abline(h=0)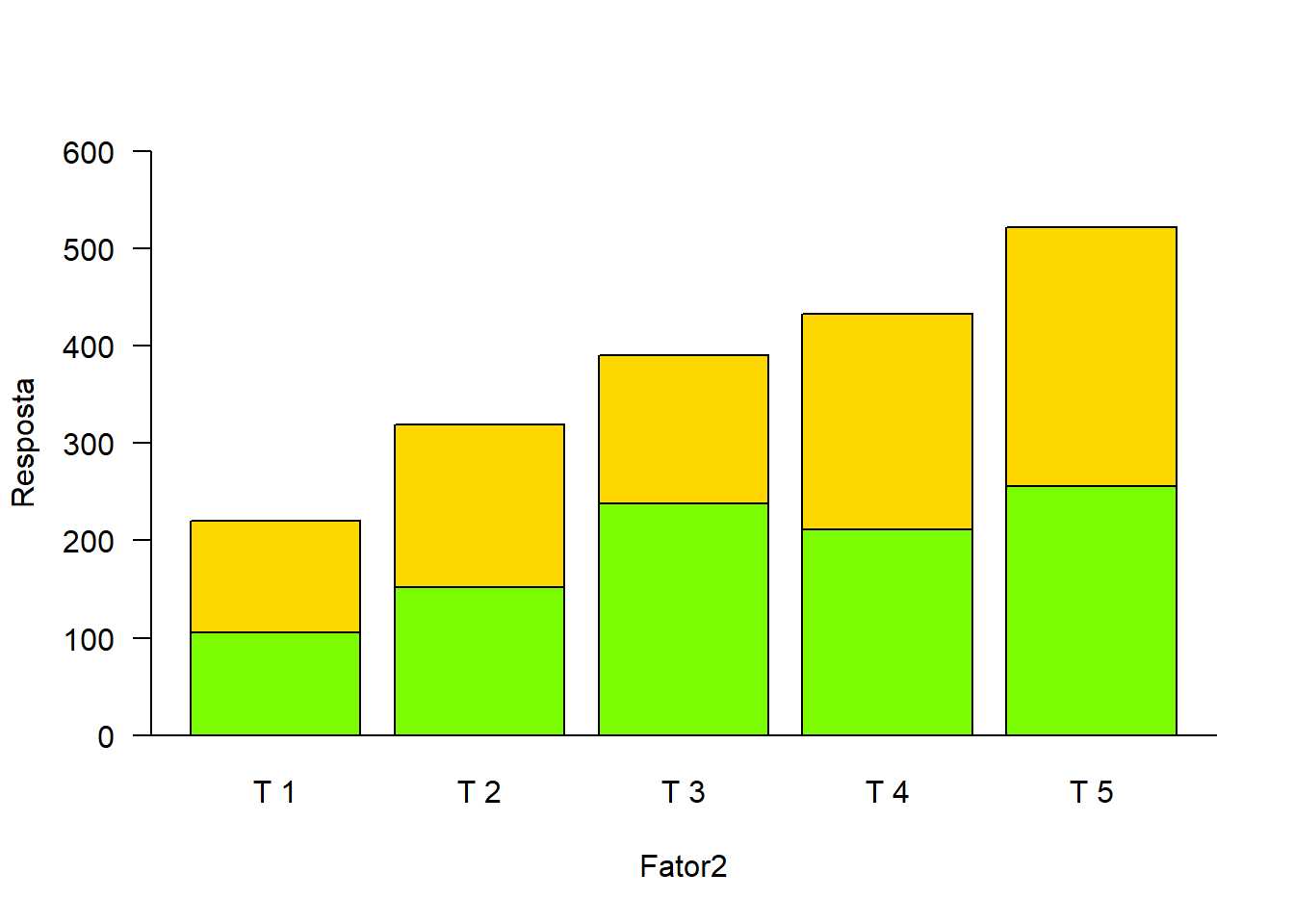
Comandos:
las=1: deixar escala do eixo Y na vertical
col=“cor”: mudar cor das barras (Ex. “red,”“blue,”“green” ou gray.colors(quantidade de tonalidades) para escala cinza ou rainbow(quantidade de cores) para escala colorida. Também é possível específicar a cor de cada barra (col=c(“red,”“green,”“yellow,”“gray,”“blue”))).
xlab e ylab: nomear eixo X e Y
xlim e ylim: escala do eixo X e Y
abline(h=0): linha na horizontal em Y=0 (No caso de vertical, abline(v=0)). É possível alterar a cor pela função “col=”cor"" e o tracejado pelo “lty=número” (Ver o Help do comando)
2.13.4 Barras de desvio-padrão
bar=barplot(media,
beside=F,
las=1,
col=c("lawngreen","gold"),
ylab="Resposta",
xlab="Tratamentos",
ylim=c(0,600))
abline(h=0)
arrows(bar,media[1,]+desvio[1,],bar,media[1,]-desvio[1,],length = 0.1,angle=90,code=3)
arrows(bar,media[1,]+media[2,]+desvio[2,],bar,media[1,]+media[2,]-desvio[2,],length = 0.1,angle=90,code=3)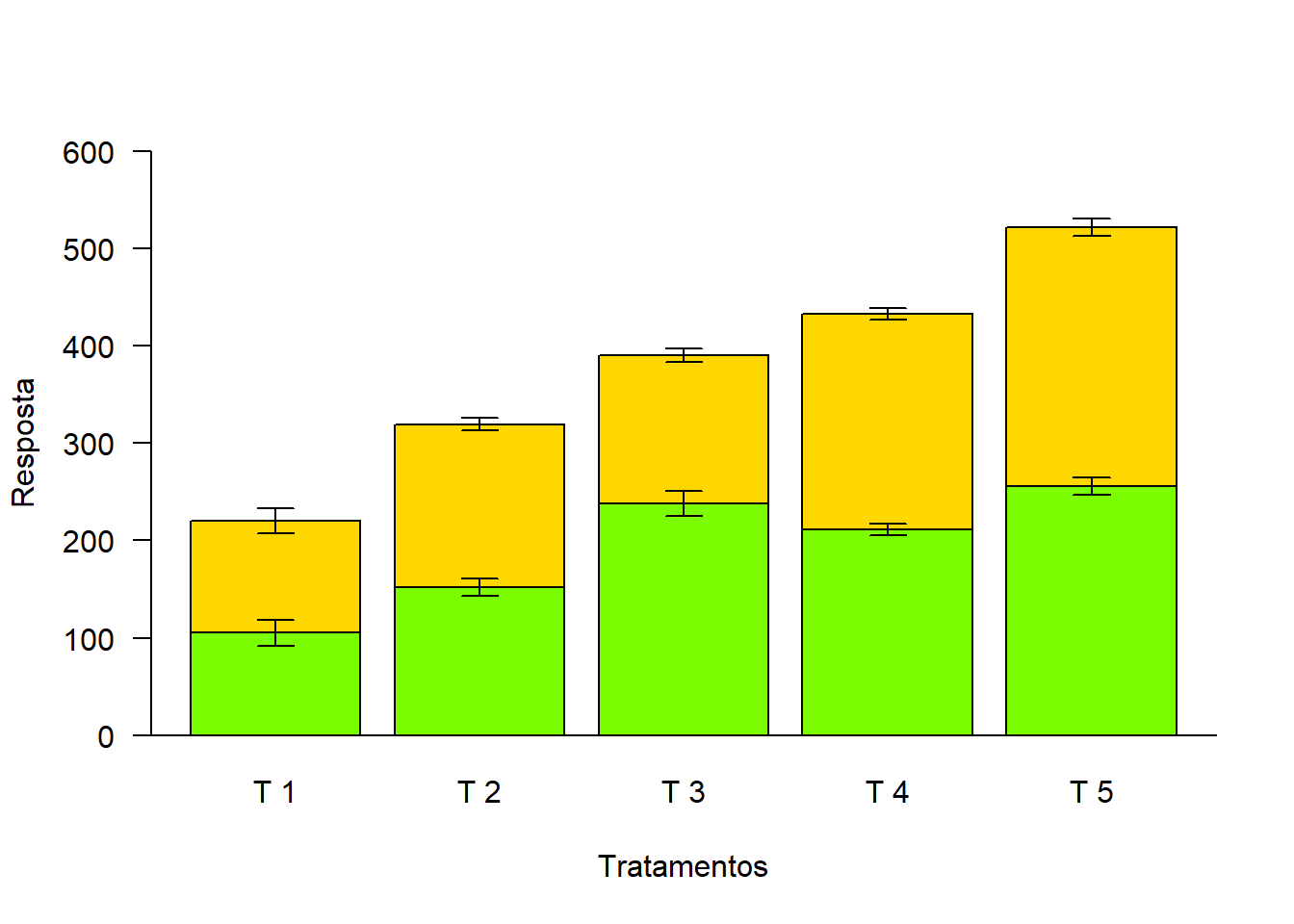
2.13.5 Unidade do eixo Y
(Ex. \(Kg\ ha^{-1}\))
bar=barplot(media, beside=F,
las=1,col=c("lawngreen","gold"),
ylab=expression(Resposta~~(kg~ha^-1)),
xlab="Tratamentos",
ylim=c(0,600))
abline(h=0)
arrows(bar,media[1,]+desvio[1,],bar,media[1,]-desvio[1,],length = 0.1,angle=90,code=3)
arrows(bar,media[1,]+media[2,]+desvio[2,],bar,media[1,]+media[2,]-desvio[2,],length = 0.1,angle=90,code=3)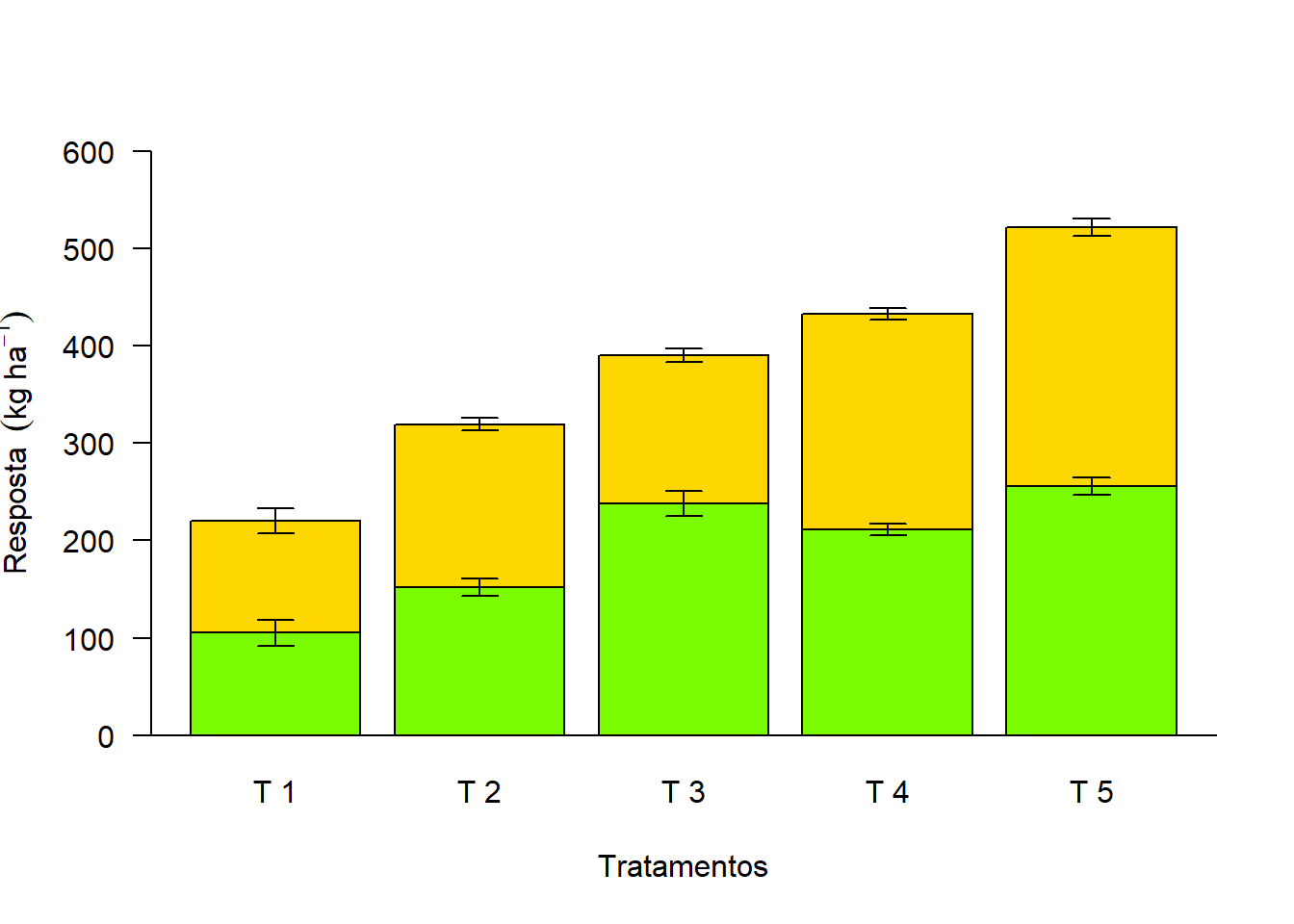
2.13.6 Média acima das barras
bar=barplot(media, beside=F,
las=1,col=c("lawngreen","gold"),
ylab=expression(Resposta~~(kg~ha^-1)),
xlab="Tratamentos",
ylim=c(0,600))
abline(h=0)
text(bar,media[1,]+desvio[1,]+20,media[1,], cex=0.8)
text(bar,media[1,]+media[2,]+desvio[2,]+20,media[2,], cex=0.8)
arrows(bar,media[1,]+desvio[1,],bar,media[1,]-desvio[1,],length = 0.1,angle=90,code=3)
arrows(bar,media[1,]+media[2,]+desvio[2,],bar,media[1,]+media[2,]-desvio[2,],length = 0.1,angle=90,code=3)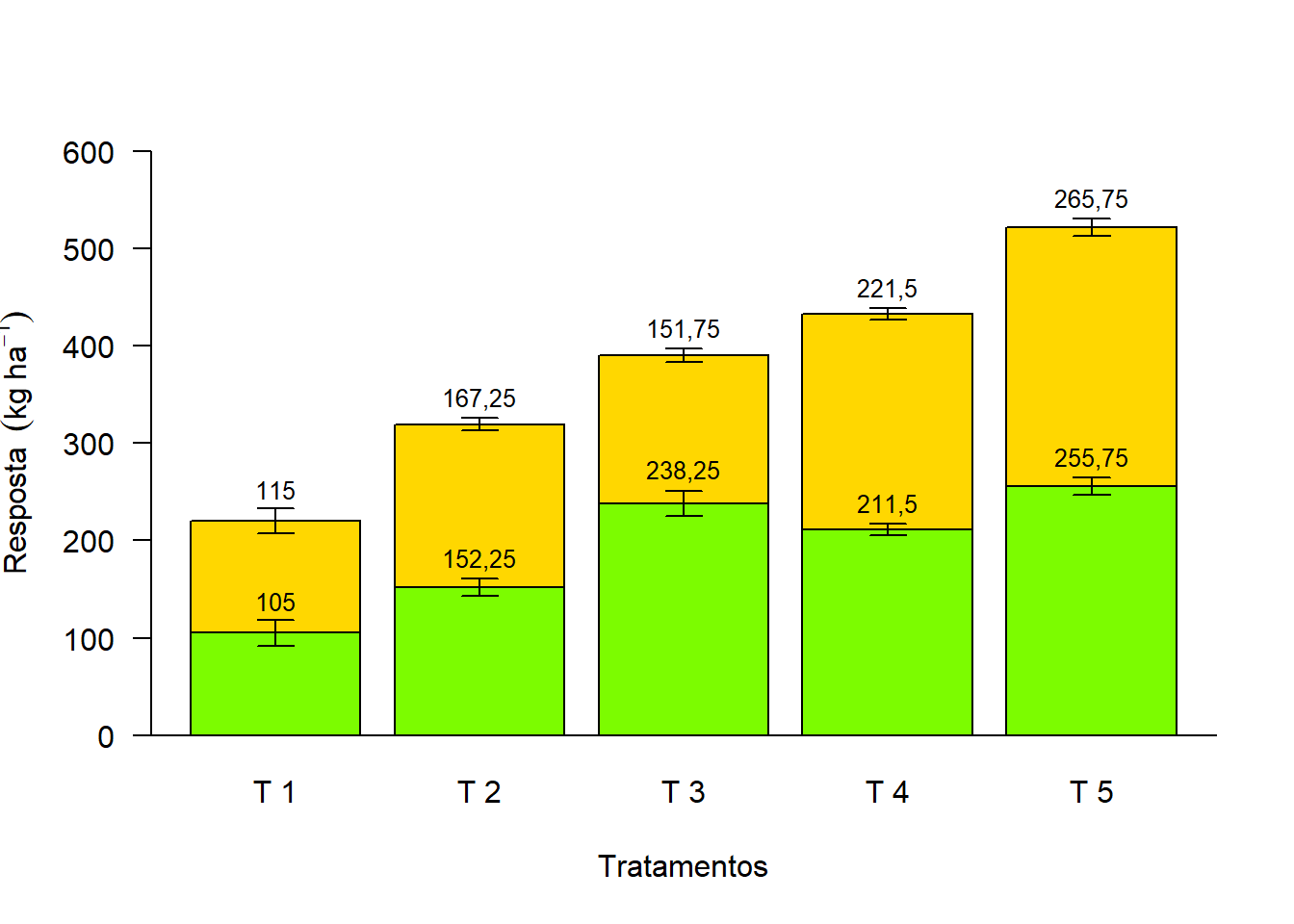
2.13.7 Separação de casa decimal
options(OutDec=",")
bar=barplot(media, beside=F,
las=1,col=c("lawngreen","gold"),
ylab=expression(Resposta~~(kg~ha^-1)),
xlab="Tratamentos",
ylim=c(0,600))
abline(h=0)
text(bar,media[1,]+desvio[1,]+20,media[1,], cex=0.8)
text(bar,media[1,]+media[2,]+desvio[2,]+20,media[2,], cex=0.8)
arrows(bar,media[1,]+desvio[1,],bar,media[1,]-desvio[1,],length = 0.1,angle=90,code=3)
arrows(bar,media[1,]+media[2,]+desvio[2,],bar,media[1,]+media[2,]-desvio[2,],length = 0.1,angle=90,code=3)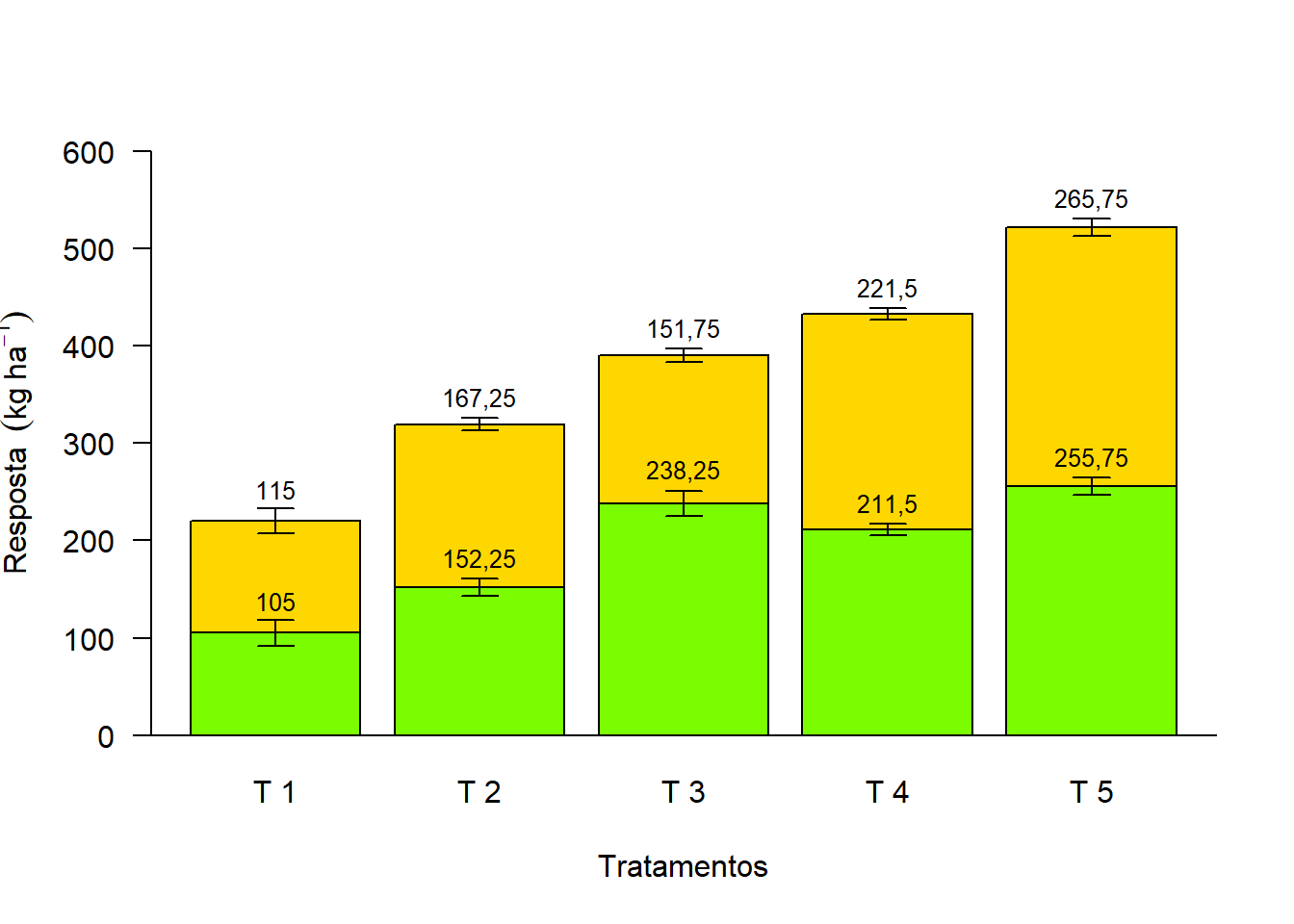
2.13.8 Letras do teste de comparação
tukey=c("dB","dA","cB","cA","cB","cA","bB","bA","aB","aA")
options(OutDec=",")
bar=barplot(media, beside=F,
las=1,col=c("lawngreen","gold"),
ylab=expression(Resposta~~(kg~ha^-1)),
xlab="Tratamentos",
ylim=c(0,600))
abline(h=0)
text(bar,media[1,]+desvio[1,]+20,paste(media[1,], tukey[c(1,3,6,7,9)]), cex=0.8)
text(bar,media[1,]+media[2,]+desvio[2,]+20,paste(media[2,], tukey[c(2,4,6,8,10)]), cex=0.8)
arrows(bar,media[1,]+desvio[1,],bar,media[1,]-desvio[1,],length = 0.1,angle=90,code=3)
arrows(bar,media[1,]+media[2,]+desvio[2,],bar,media[1,]+media[2,]-desvio[2,],length = 0.1,angle=90,code=3)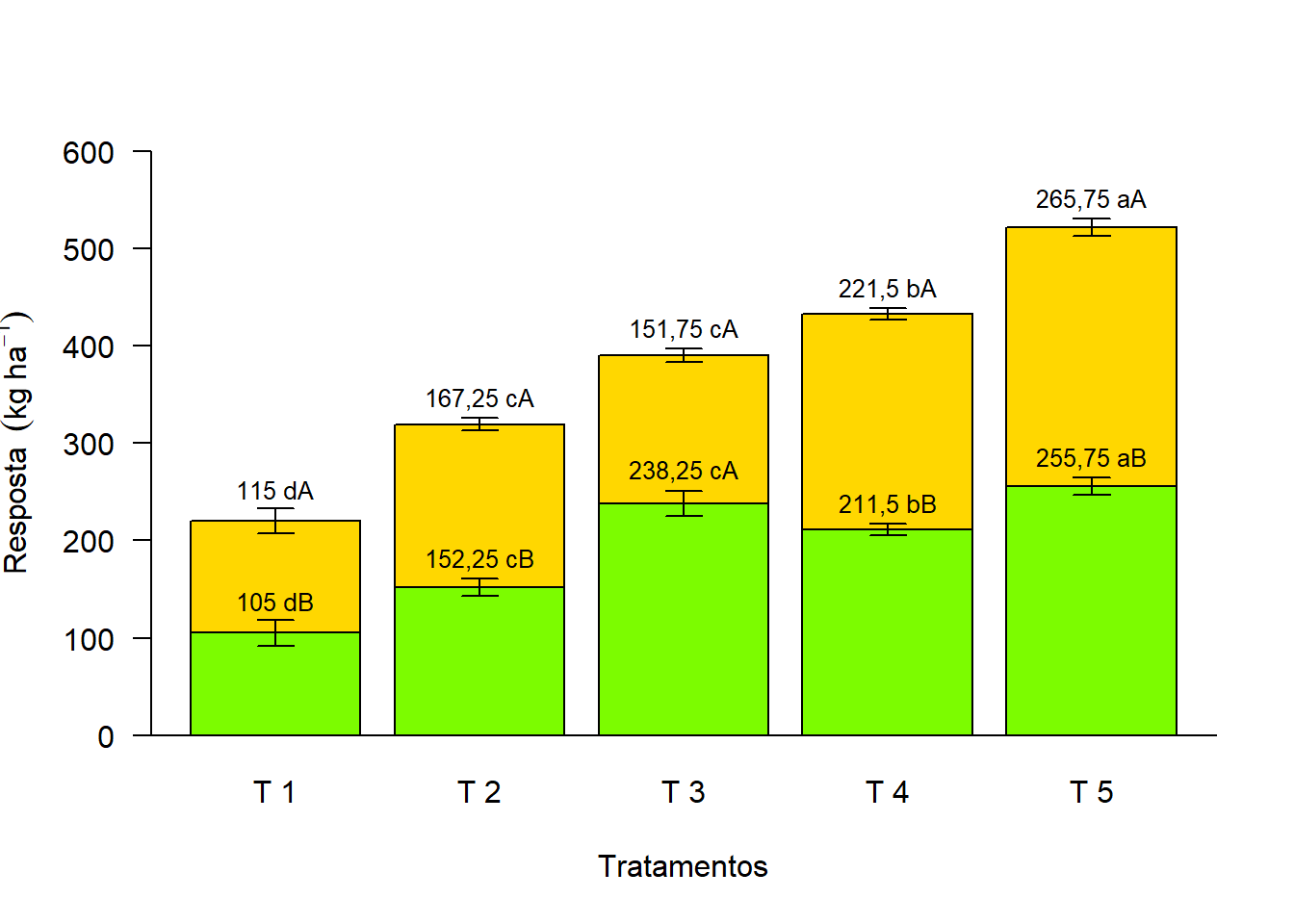
2.13.9 Adicionando legenda
legend.text=rownames(media): adicionar a legenda (neste caso em relação ao Fator 2)
args.legend: argumentos da legenda (x=“topleft”: legenda será adicionada no parte superior esquerda, podemos adicionar superior direito (“topright”), inferior esquerdo (“bottomleft”), inferior direito (“bottomright”), centralizado (“center”))
tukey=c("dB","dA","cB","cA","cB","cA","bB","bA","aB","aA")
options(OutDec=",")
bar=barplot(media,
beside=F,
legend.text = rownames(media),
args.legend = list(x="topleft", bty="n"),
las=1,col=c("lawngreen","gold"),
ylab=expression(Resposta~(kg~ha^-1)),
xlab="Tratamentos",
ylim=c(0,600))
abline(h=0)
text(bar,media[1,]+desvio[1,]+20,paste(media[1,], tukey[c(1,3,6,7,9)]), cex=0.8)
text(bar,media[1,]+media[2,]+desvio[2,]+20,paste(media[2,], tukey[c(2,4,6,8,10)]), cex=0.8)
arrows(bar,media[1,]+desvio[1,],bar,media[1,]-desvio[1,],length = 0.1,angle=90,code=3)
arrows(bar,media[1,]+media[2,]+desvio[2,],bar,media[1,]+media[2,]-desvio[2,],length = 0.1,angle=90,code=3)
2.14 Dois lados com escala positiva
2.14.1 Conjunto de dados
trat=rep(c("T1","T2","T3","T4","T5"),e=3)
mspa=c(8,10,12,18,20,22,28,30,32,38,40,42,48,50,52)
msr=c(14,15,16,19,20,21,24,25,26,29,30,31,34,35,36)2.14.2 Média e desvio-padrão
m1=tapply(mspa, trat, mean)
m2=tapply(msr, trat, mean)
sd1=tapply(mspa, trat, sd)
sd2=tapply(msr, trat, sd)# alterando margem e configurando para dois plots um abaixo do outro
op <- list(mfrow = c(2,1),
oma = c(5,4,0,0) + 0.1,
mar = c(0,0,0,1))2.14.3 Somente colunas
Obs. Nesse caso em específico, estamos querendo que ambas as variáveis assumem respostas positivas. Todavia, queremo a coluna da variável MSPA acima e MSR abaixo.
par(op)
b1=barplot(m1,
axes=F,
col="blue",
ylim=c(0,60),
axisnames = F,
las=1)
b2=barplot(m2,
axes=F,
col="red",
ylim=c(60,0),
las=1)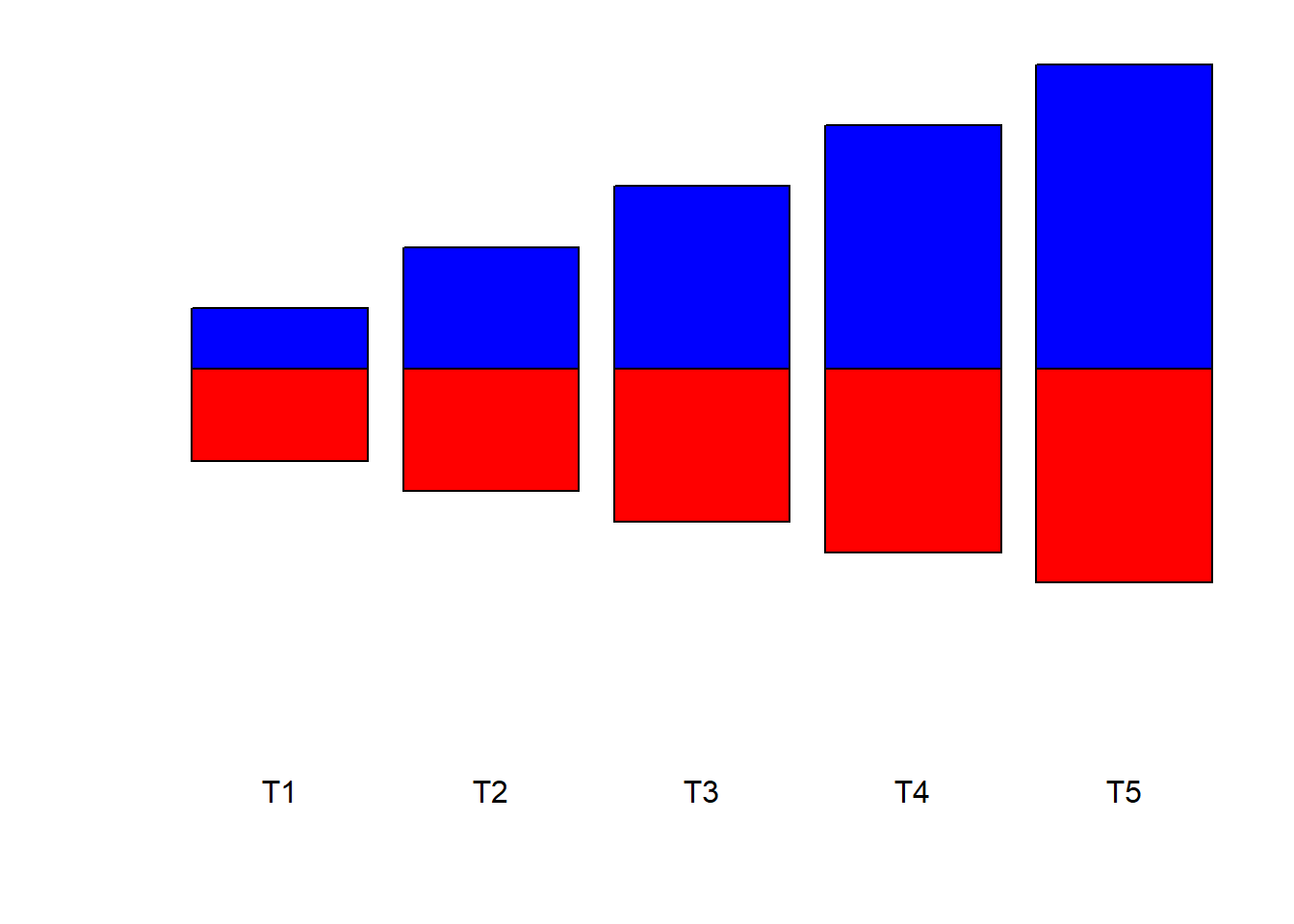
2.14.4 Escala do eixo Y
par(op)
b1=barplot(m1,
axes=F,
col="blue",
ylim=c(0,60),
axisnames = F,
las=1)
axis(2,seq(0,50,10),las=1)
b1=barplot(m2,
axes=F,
col="red",
ylim=c(60,0),
axisnames = F,
las=1)
axis(2,seq(0,50,10),las=1)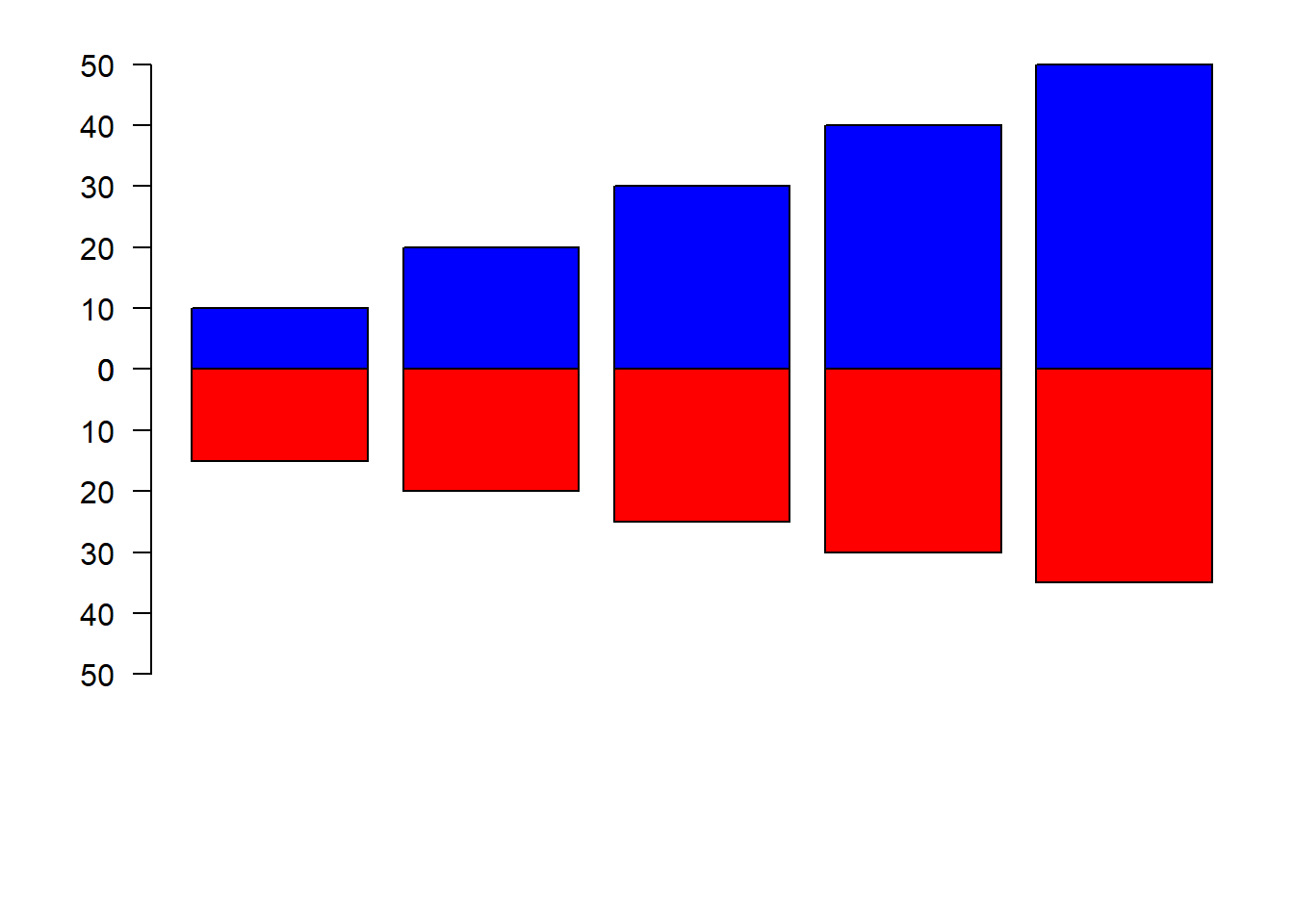
2.14.5 Barras de desvio-padrão
par(op)
b1=barplot(m1,
axes=F,
col="blue",
ylim=c(0,60),
axisnames = F,
las=1)
axis(2,seq(0,50,10),las=1)
arrows(b1,m1+sd1,b1,m1-sd1,angle = 90,code=3, length = 0.05)
b2=barplot(m2,
axes=F,
col="red",
ylim=c(60,0),
axisnames = F,
las=1)
axis(2,seq(0,50,10),las=1)
arrows(b2,m2+sd2,b2,m2-sd2,angle = 90,code=3, length = 0.05)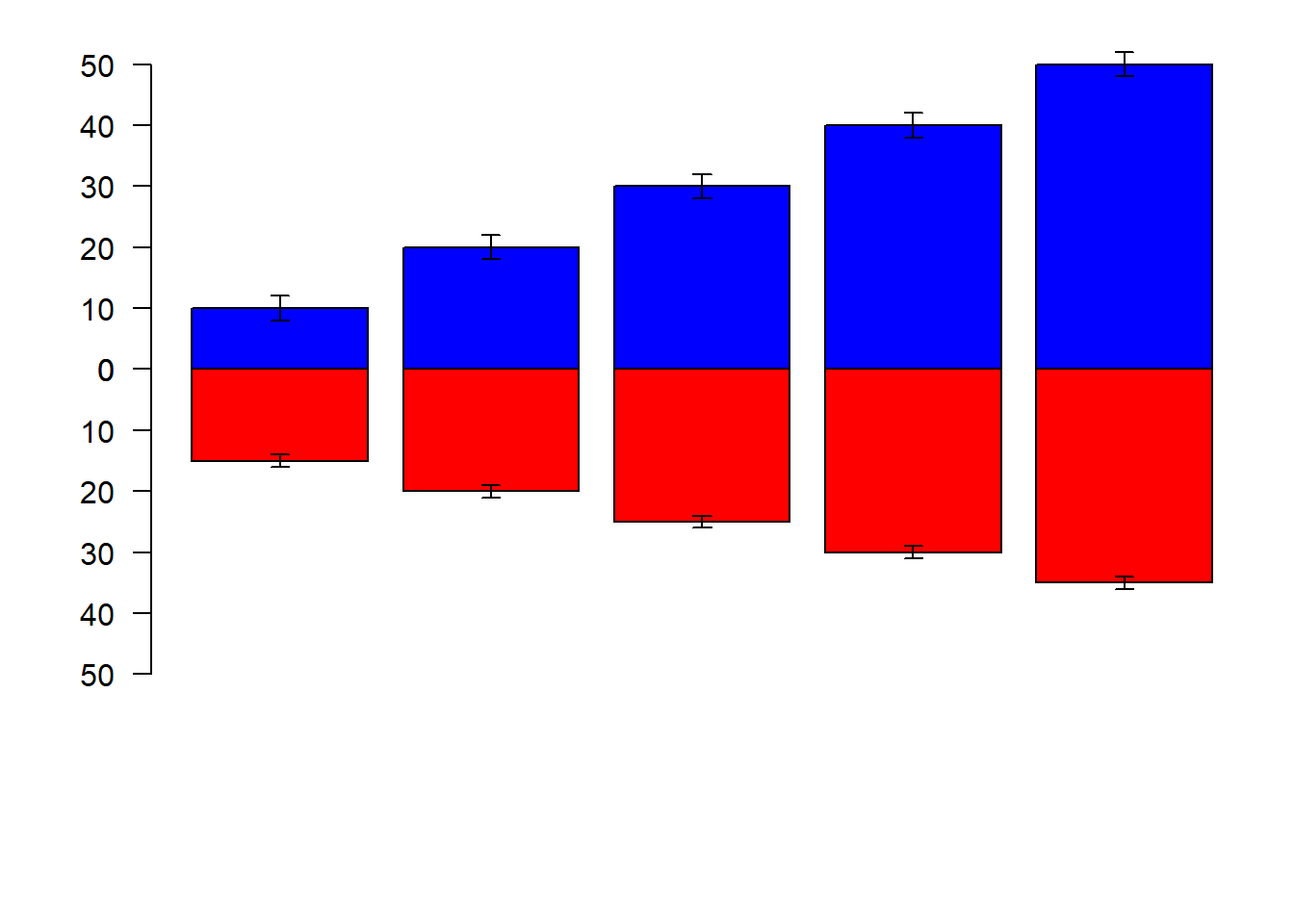
2.14.6 Linha em 0 e título de Y
par(op)
b1=barplot(m1,
axes=F,
col="blue",
ylim=c(0,60),
axisnames = F,
las=1)
axis(2,seq(0,50,10),las=1)
arrows(b1,m1+sd1,b1,m1-sd1,angle = 90,code=3, length = 0.05)
b2=barplot(m2,
axes=F,
col="red",
ylim=c(60,0),
axisnames = F,
las=1)
axis(2,seq(0,50,10),las=1)
title(ylab = "Resposta",outer=T, line = 3)
arrows(b2,m2+sd2,b2,m2-sd2,angle = 90,code=3, length = 0.05)
abline(h=0)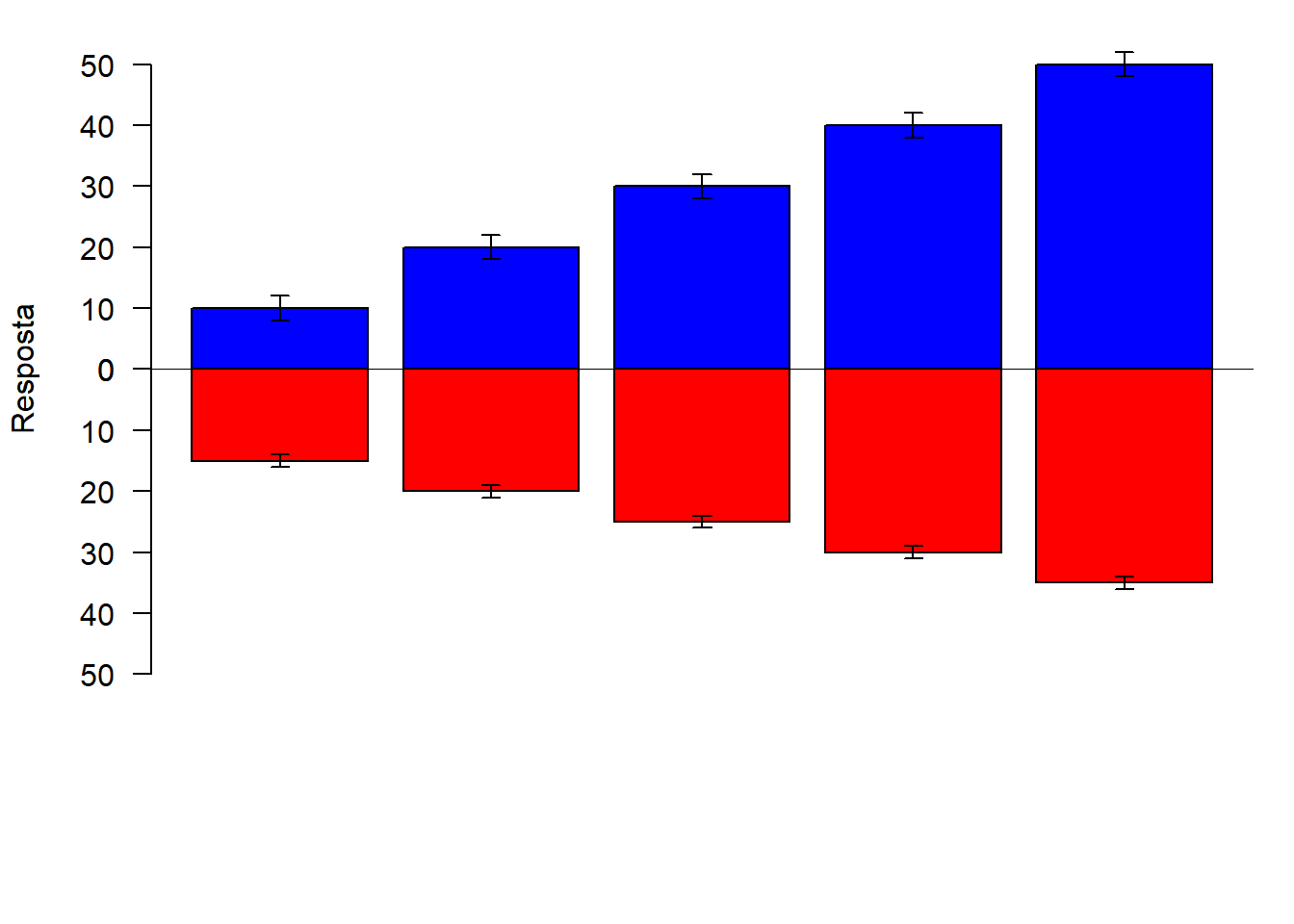
2.14.7 Título para MS (g)
par(op)
b1=barplot(m1,
axes=F,
col="blue",
ylim=c(0,60),
axisnames = F,
las=1)
axis(2,seq(0,50,10),las=1)
arrows(b1,m1+sd1,b1,m1-sd1,angle = 90,code=3, length = 0.05)
b2=barplot(m2,
axes=F,
col="red",
ylim=c(60,0),
las=1)
axis(2,seq(0,50,10),las=1)
title(ylab = expression(MS~(g)),outer=T, line = 3)
arrows(b2,m2+sd2,b2,m2-sd2,angle = 90,code=3, length = 0.05)
abline(h=0)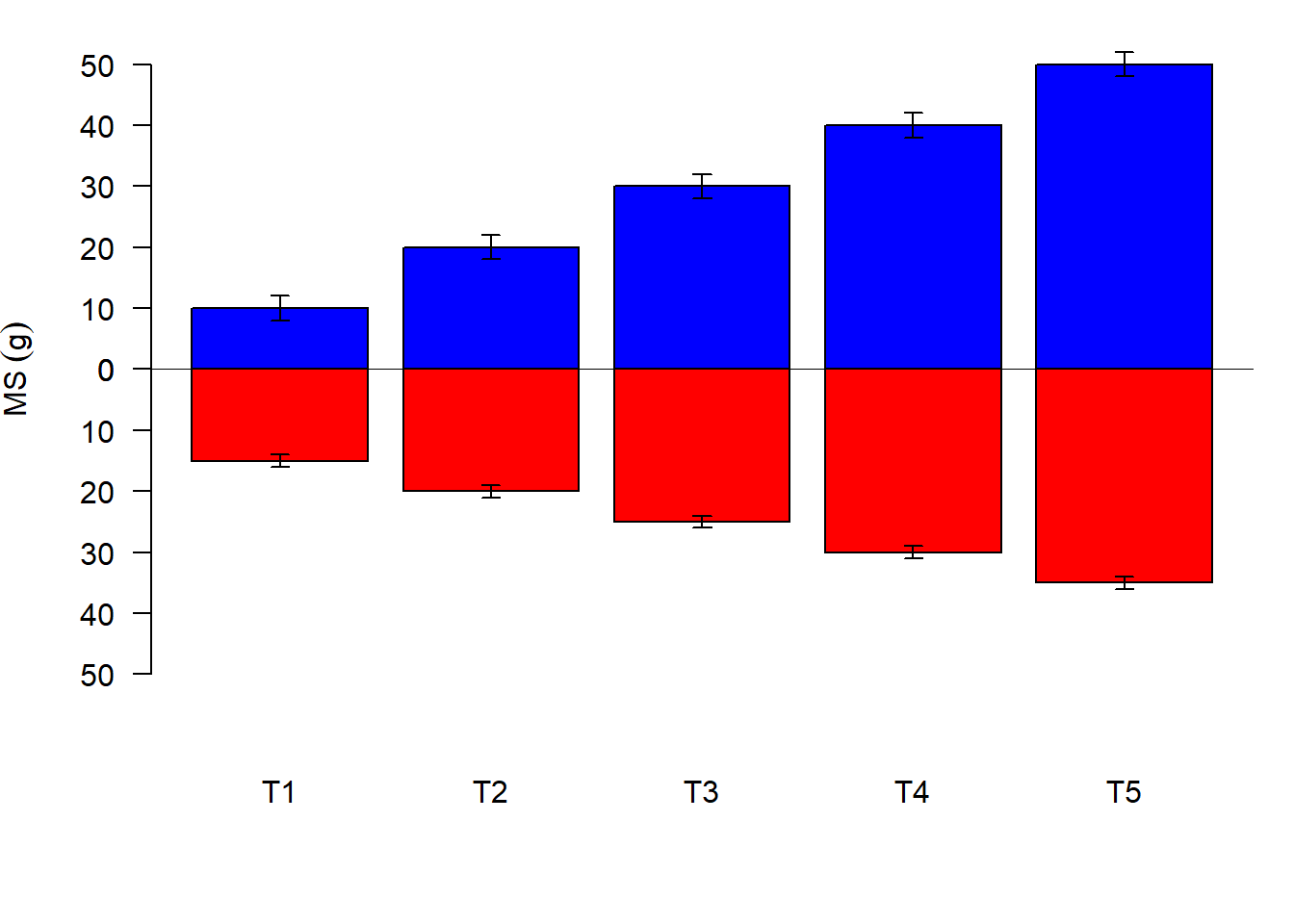
2.14.8 Coluna hachurada
par(op)
b1=barplot(m1,
axes=F,
col="blue",
density = 40,
ylim=c(0,60),
axisnames = F,
las=1)
axis(2,seq(0,50,10),las=1)
arrows(b1,m1+sd1,b1,m1-sd1,angle = 90,code=3, length = 0.05)
b2=barplot(m2,
axes=F,
col="red",
ylim=c(60,0),
density = 20,
las=1)
axis(2,seq(0,50,10),las=1)
title(ylab = expression(MS~(g)),outer=T, line = 3)
arrows(b2,m2+sd2,b2,m2-sd2,angle = 90,code=3, length = 0.05)
abline(h=0)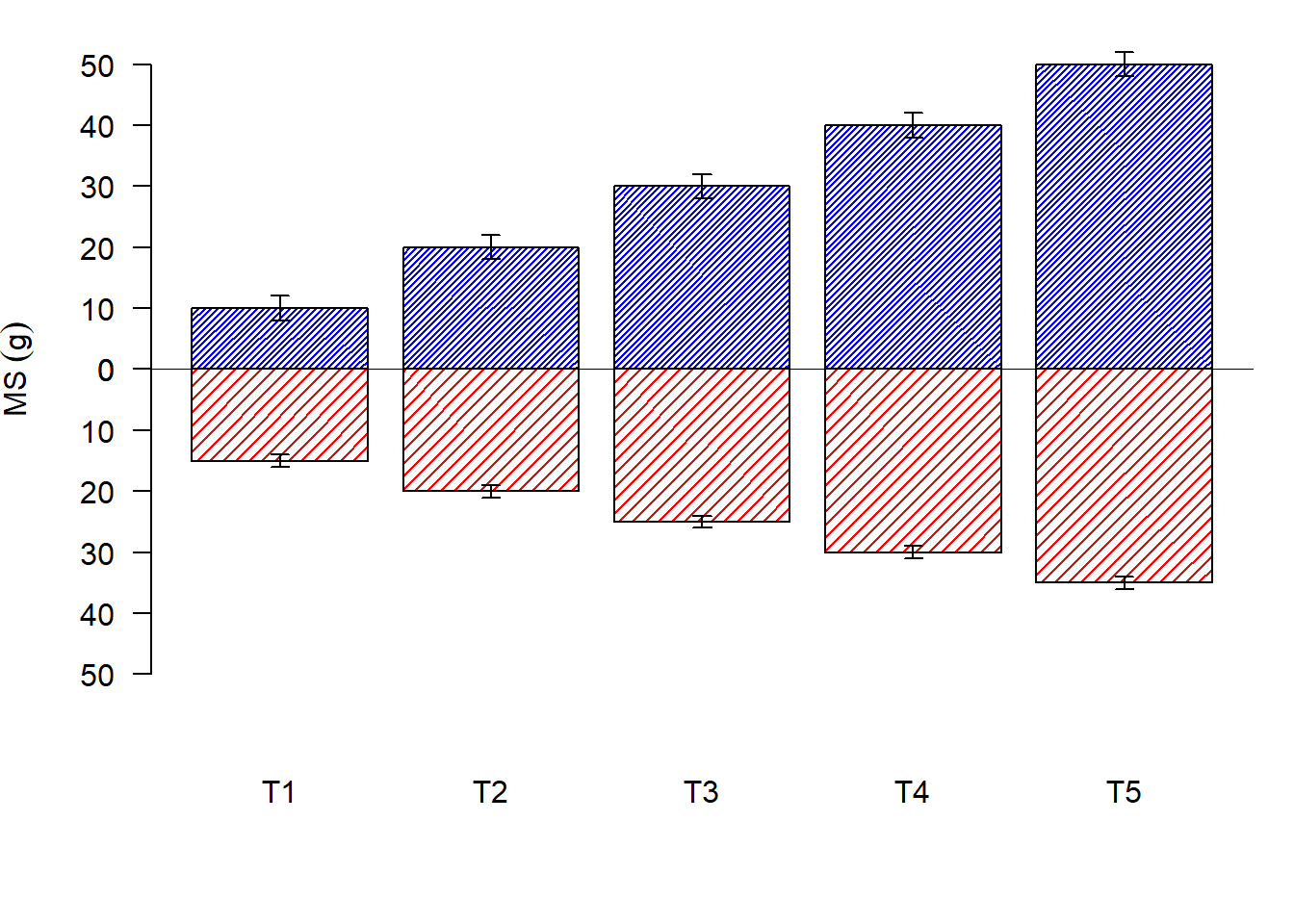
2.14.9 Adicionando legenda
par(op)
b1=barplot(m1,
axes=F,
col="blue",
density = 40,
ylim=c(0,60),
axisnames = F,
las=1)
axis(2,seq(0,50,10),las=1)
arrows(b1,m1+sd1,b1,m1-sd1,angle = 90,code=3, length = 0.05)
legend("topleft",
fill=c("blue","red"),
legend=c("MSPA","MSR"),
density = c(40,20),
bty="n")
b2=barplot(m2,
axes=F,
col="red",
ylim=c(60,0),
density = 20,
las=1)
axis(2,seq(0,50,10),las=1)
title(ylab = expression(MS~(g)),outer=T, line = 3)
arrows(b2,m2+sd2,b2,m2-sd2,angle = 90,code=3, length = 0.05)
abline(h=0)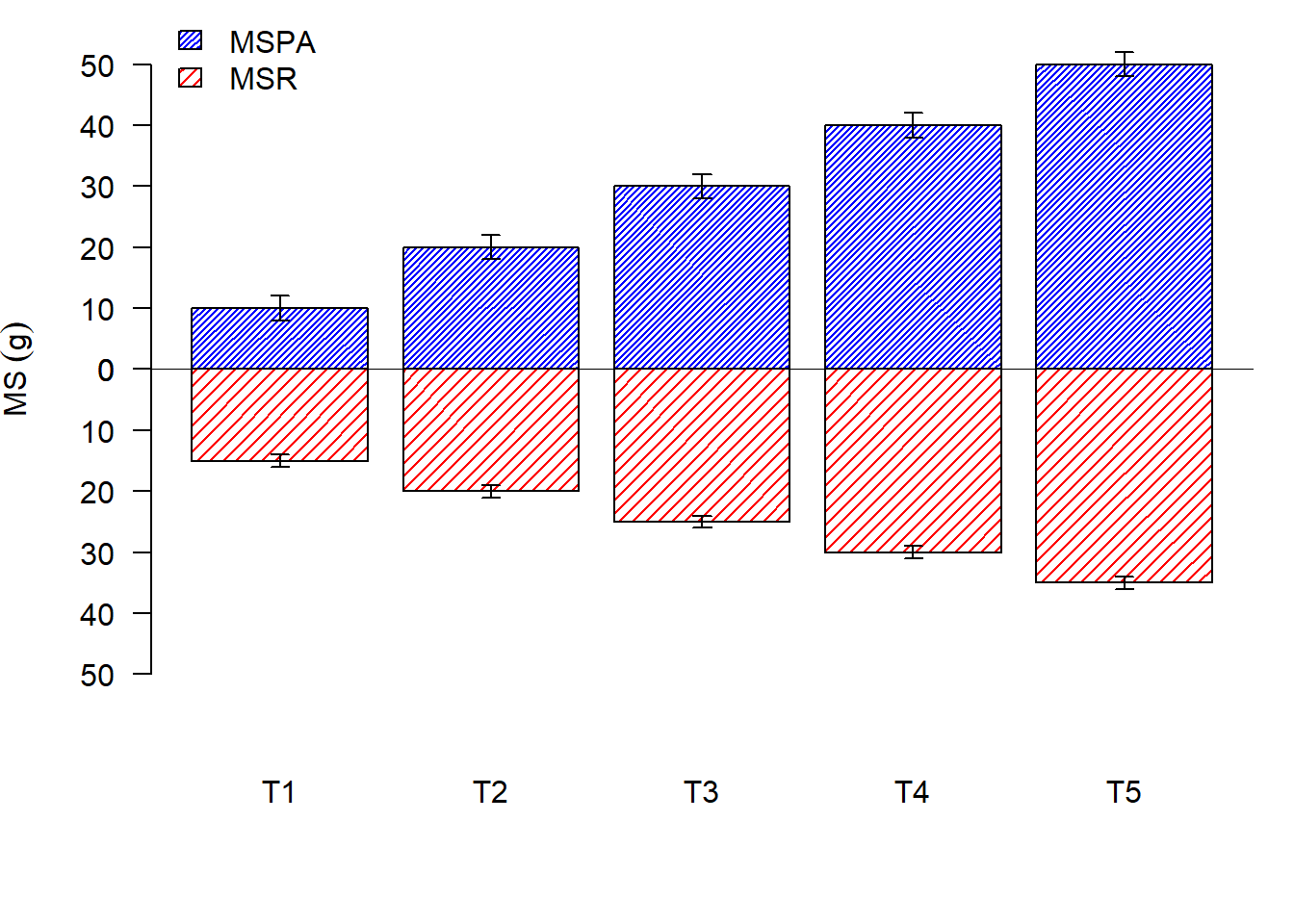
2.14.10 Teste de comparação
par(op)
b1=barplot(m1,
axes=F,
col="blue",
density = 40,
ylim=c(0,60),
axisnames = F,
las=1)
axis(2,seq(0,50,10),las=1)
arrows(b1,m1+sd1,b1,m1-sd1,angle = 90,code=3, length = 0.05)
legend("topleft",
fill=c("blue","red"),
legend=c("MSPA","MSR"),
density = c(40,20),
bty="n")
text(b1,m1+sd1+5,c("e","d","c","b","a"))
b2=barplot(m2,
axes=F,
col="red",
ylim=c(60,0),
density = 20,
las=1)
axis(2,seq(0,50,10),las=1)
text(b2,m2+sd2+5,c("e","d","c","b","a"))
title(ylab = expression(MS~(g)),outer=T, line = 3)
arrows(b2,m2+sd2,b2,m2-sd2,angle = 90,code=3, length = 0.05)
abline(h=0)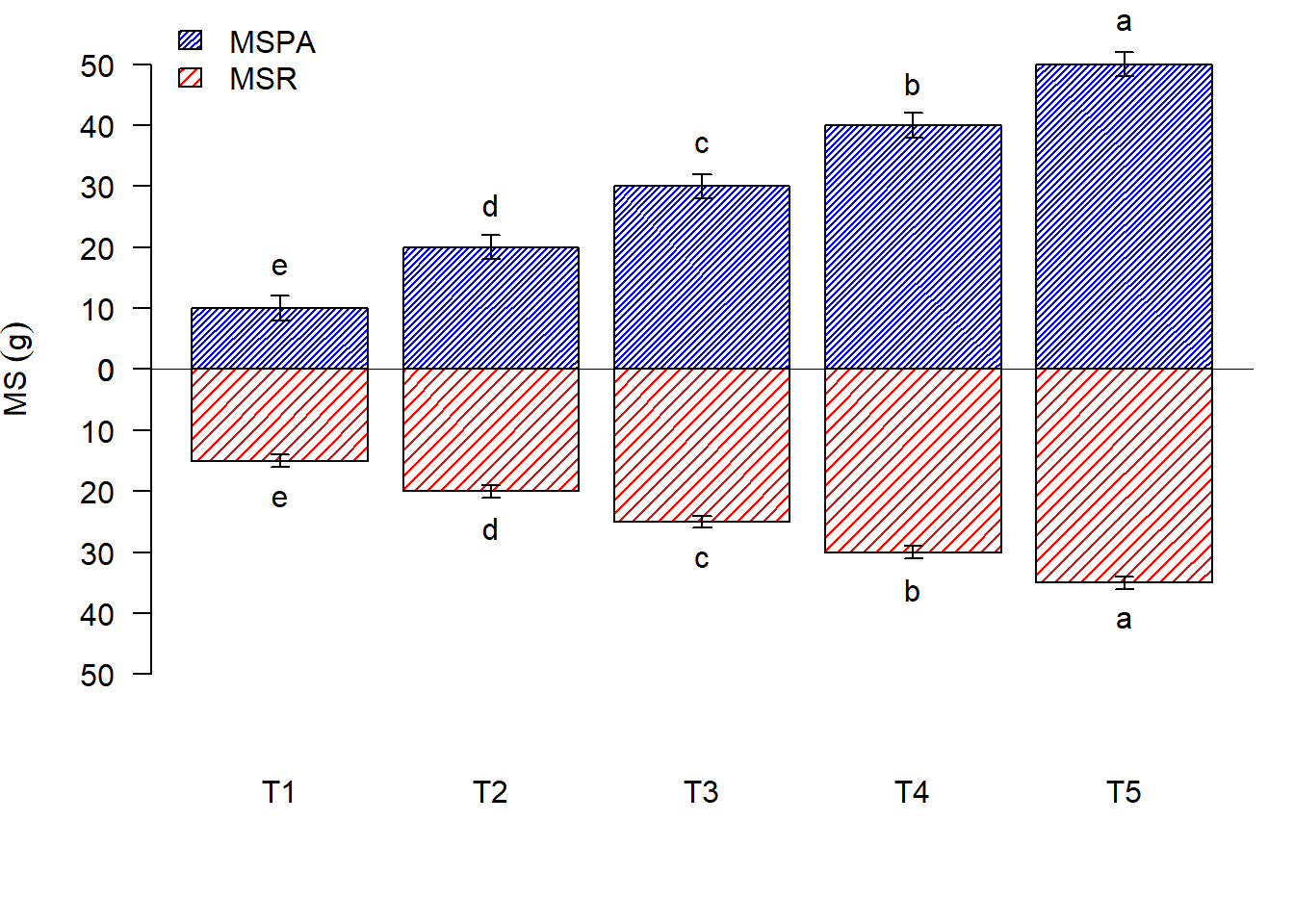
2.14.11 Mudando fonte
par(family="serif")
par(op)
b1=barplot(m1,
axes=F,
col="blue",
density = 40,
ylim=c(0,60),
axisnames = F,
las=1)
axis(2,seq(0,50,10),las=1)
arrows(b1,m1+sd1,b1,m1-sd1,angle = 90,code=3, length = 0.05)
legend("topleft",
fill=c("blue","red"),
legend=c("MSPA","MSR"),
density = c(40,20),
bty="n")
text(b1,m1+sd1+5,c("e","d","c","b","a"))
b2=barplot(m2,
axes=F,
col="red",
ylim=c(60,0),
density = 20,
las=1)
axis(2,seq(0,50,10),las=1)
text(b2,m2+sd2+5,c("e","d","c","b","a"))
title(ylab = expression(MS~(g)),outer=T, line = 3)
arrows(b2,m2+sd2,b2,m2-sd2,angle = 90,code=3, length = 0.05)
abline(h=0)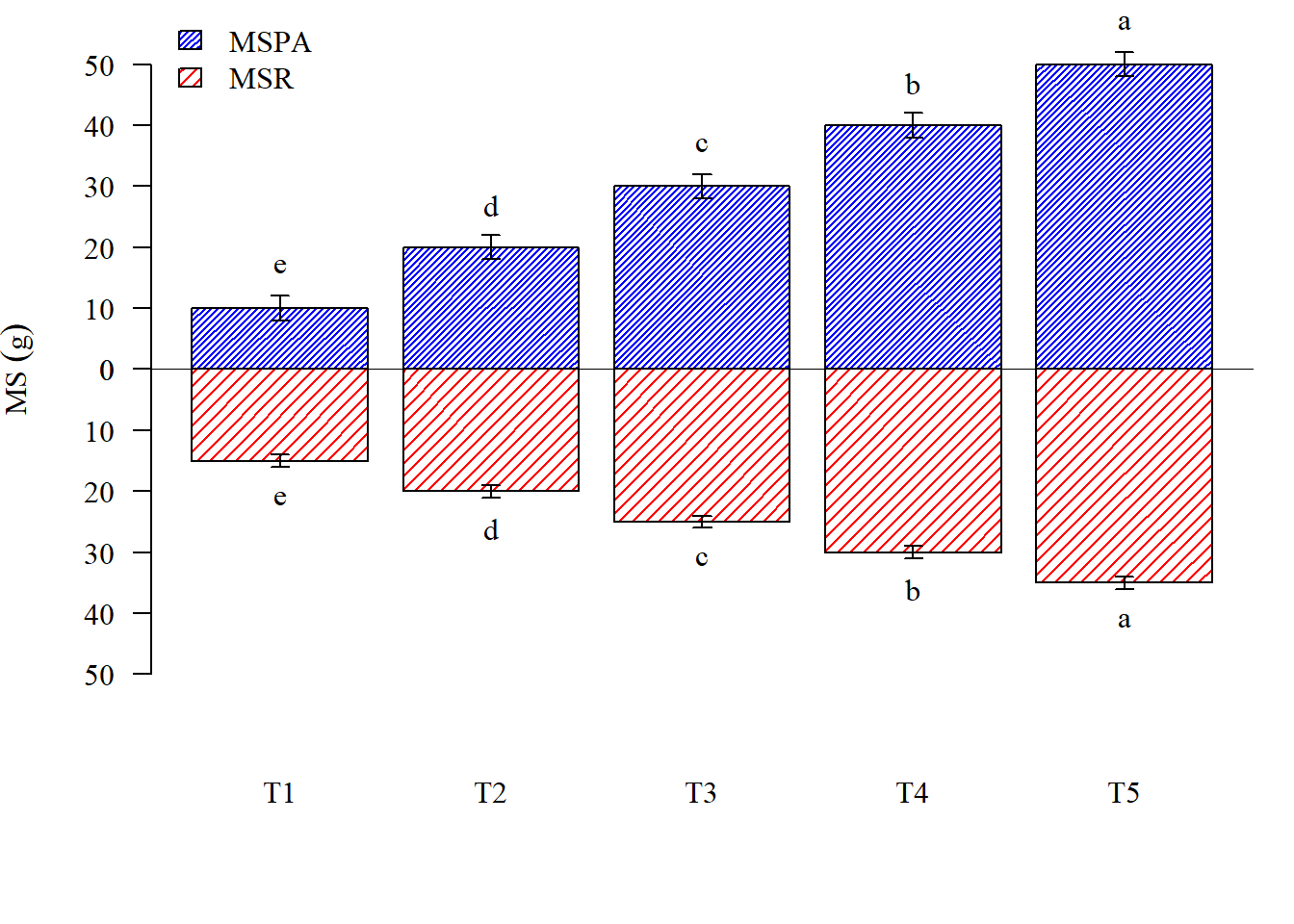 ****
****
2.15 Dois lados com escala positiva (ggplot2)
2.15.1 Conjunto de dados
trat=rep(c("T1","T2","T3","T4","T5"),e=3)
mspa=c(8,10,12,18,20,22,28,30,32,38,40,42,48,50,52)
msr=c(14,15,16,19,20,21,24,25,26,29,30,31,34,35,36)2.15.2 Média e desvio-padrão
m1=tapply(mspa, trat, mean)
m2=tapply(msr, trat, mean)
d1=tapply(mspa, trat, sd)
d2=tapply(msr, trat, sd)
dados=data.frame(trat=rep(names(m1),2),
resp=c(m1,-m2),
pos=c(m1+d1,-m2-d2),
comp=rep(c("MSPA","MSR"),e=5),
letra=c("e","d","c","b","a",
"e","d","c","b","a"))library(ggplot2)
ggplot(dados,aes(x=trat,y=resp,fill=comp))+
geom_col(color="black")+
scale_y_continuous(expand = c(0, 1),
breaks = pretty(dados$resp),
labels = abs(pretty(dados$resp)))+
theme_bw()+xlab("")+ylab("Massa seca (g)")+
geom_text(aes(y=pos+c(rep(c(0.3,-0.3),e=5)),
label=paste(abs(resp),letra)))